文章信息
- 孙丽冉, 齐迎菊, 田晓轩
- SUN Liran, QI Yingju, TIAN Xiaoxuan
- 哈氏蜈蚣线粒体基因组全序列分析
- Analysis of mitochondrial genome of Scolopendra subspinipes dehaani
- 天津中医药, 2018, 35(3): 225-229
- Tianjin Journal of Traditional Chinese Medicine, 2018, 35(3): 225-229
- http://dx.doi.org/10.11656/j.issn.1672-1519.2018.03.19
-
文章历史
- 收稿日期: 2017-12-22
2. 天津国际生物医药联合研究院中药新药研发中心, 天津 300457
蜈蚣是多足亚门(Myriapoda)唇足纲(Chilopoda)最大最古老的陆生节肢动物,大约有3 300~3 500种蜈蚣分布在除南极洲以外的每个洲[1]。蜈蚣主要以昆虫、蚯蚓、蜗牛甚至小脊椎动物为食;因为缺乏蜡质的昆虫表皮,容易失水,通常栖息于森林的叶、树皮、木材和土壤中[2]。蜈蚣的第一对四肢从身体向前伸展覆盖口,四肢末端有锋利的爪子和毒腺。毒腺分泌的毒液不仅可以杀死猎物,也可防御天敌,保护自己的安全。被毒液伤害的人会有局部疼痛,发红,肿胀,表面坏死,寒战,发热,虚弱等症状,严重的继发感染甚至会导致人的死亡[3-6]。
蜈蚣作为传统中药,主治半身不遂、破伤风、小儿惊风、中风等病[7],其药效物质主要是脂肪酸、蛋白质、酶及胆甾醇等物质[8]。其中,神经毒素、离子通道作用的成分及毒液过敏原是哈氏蜈蚣毒液的主要成分。纯化的哈氏蜈蚣毒液蛋白或多肽具有血小板聚集活性、抗凝活性磷脂酶A2活性、胰蛋白酶抑制活性、电压门控钾通道活性等药理特性[9]。现代药理学研究发现,蜈蚣可调节机体脂代谢,降低血脂,保护心肌细胞及血管内皮细胞免受脂质过氧化及缺血再灌损伤,增加冠脉血流量。同时,蜈蚣能够提高人体免疫力,具有改善机体免疫功能和抗衰老功能。除此以外,蜈蚣水提液可增强肠胃功能,且具有一定的镇静、镇痛作用。体外实验结果表明,蜈蚣水提取物对肝癌、肾癌、肺癌、胃癌、结肠癌、卵巢癌等多种癌症细胞有显著的生长抑制作用[10-14]。
中国供药用的蜈蚣除药典收载的少棘巨蜈蚣外,还有哈氏蜈蚣、墨江蜈蚣、多棘蜈蚣等6个物种在中国不同地区作药用蜈蚣使用。哈氏蜈蚣(Scolopendra subspinipes dehaani)在分类上属于蜈蚣目(Scolopendromorpha)蜈蚣科(Scolopendridae),是一种体形较长的(通常为6~20 cm)、毒液毒性较大的蜈蚣[15-16]。哈氏蜈蚣主要分布在广东、广西、海南及云南西双版纳等南部热带地区,数量较少。不同蜈蚣品种间所含药效物质及毒性物质不尽相同,对蜈蚣品种的准确鉴定可保证用药安全,而线粒体基因组序列可为其鉴定提供依据。
动物线粒体基因组中含有从分子序列到基因结构的所有信息,其基因的组成排列相对保守,可以对许多特征进行综合比较[17]。且与核基因组相比,线粒体基因组相对较小,易于分析,利于进行基于基因组的进化与生物类群系统发生的研究[18],被广泛应用于动物的系统发育以及种群保护等领域的研究[19]。本文采用Illumina HiSeq X Ten测序技术,获得了哈氏蜈蚣线粒体基因组的完整序列,是国内外首个哈氏蜈蚣线粒体基因组的公开报道。
1 实验材料 1.1 实验样品哈氏蜈蚣样品购于广东佛山,并经形态学鉴定和COI基因分子鉴定确定购买样品为该物种。Voucher ID为hlwg-1。
1.2 实验方法 1.2.1 线粒体DNA富集取新鲜肌肉组织立即使用。利用差速离心的方法富集线粒体细胞器,然后采用酚氯仿抽提法,获得哈氏蜈蚣总基因组DNA,并使用Nanodrop 2000粗略测定DNA浓度及纯度, 然后利用REPLI-g(R)Mitochondrial DNA Kit试剂盒对总DNA进行滚环扩增,提升线粒体DNA相对于基因组DNA的比例,得到更多线粒体DNA。
1.2.2 Illumina HiSeq X Ten高通量测序 1.2.2.1 建库利用超声波将滚环扩增产物打断为特定长度的DNA片段,并在这些片段两端连接上序列已知的通用接头构建出文库。
1.2.2.2 Flowcell构建好的文库通过flowcell时会随机附着在flowcell表面的channel上,每个channel表面有很多和建库过程中连接在DNA片段两端的接头互补配对的接头。
1.2.2.3 桥式扩增桥式PCR以flowcell表面固定的DNA链为模板,进行桥式扩增,经过变性、退火、延伸的不断循环,最终每个DNA片段都在各自的位置上形成簇。
1.2.2.4 上机测序向反应体系中同时添加包含DNA聚合酶、接头引物及带有保护基团和特异荧光的4种dNTP的混合物。在测序过程中,当dNTP被结合到合成链上之后,所有其他未使用的dNTP及DNA聚合酶都被冲洗掉,再加入所需的缓冲液,激发荧光,记录荧光信号并转化为碱基信号,然后加入化学试剂淬灭荧光基团及去掉保护基团以便进行下一轮的测序反应。
1.2.3 序列拼接将测序的原始数据从测序服务器上下载至本地,将低质量序列、过短序列以及错误序列过滤掉得到用于下游分析的序列,根据目的基因组大小,调整合适的覆盖倍数,使用MIRA 4.0等软件进行de novo拼接,得到片段较长的contigs,再结合近缘物种的序列进行mapping组装,取得质量较好的consensus替代近源物种序列,继续进行mapping组装,经反复迭代后,进行最高质量碱基合意,合意后得到完整线粒体全基因组序列。
1.2.4 注释分析和提交通过与近缘种序列进行比较以及MITOS server线粒体在线注释系统[20],对全序列进行注释。以盲蜈蚣科Scolopocryptops sp.(GenBank KC200076)的线粒体基因组全序列为参照,对蛋白质编码基因、tRNA、rRNA及控制区等进行注释,tRNA二级结构由MITOS[12]折叠生成。使用软件MEGA 4.0及Bioedit等统计哈氏蜈蚣线粒体基因组的序列总长、碱基组成以及GC含量等。完整的哈氏蜈蚣线粒体基因组使用Sequin对各基因进行详细注释后,提交到GenBank,登录号为KY947341。
2 实验结果 2.1 序列拼接结果组装得到哈氏蜈蚣线粒体基因组后,经bowtie2比对[21],确认共2 557 168条序列(reads)被用于拼接该基因组,这些序列的平均长度为150 bp,测序深度达39 293倍。
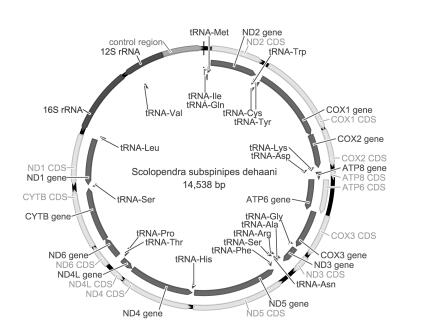
2.2 整体结构哈氏蜈蚣线粒体基因组结构见于图 1与表 1。哈氏蜈蚣共编码35个基因,包括了20个tRNA,13个蛋白编码基因,2个rRNA(16SrRNA、12SrRNA)并有1个控制区。其中除ND5、ND1、ND4、ND4L、16S rRNA、12S rRNA和8个tRNA(tRNA-Leu、tRNA-Val、tRNA-Gln、tRNA-Pro、tRNA-Cys、tRNA-Phe、tRNA-Tyr和tRNA-His)由轻链(L链)编码,其余21个基因均由重链(H链)编码。
|
| 图 1 哈氏蜈蚣线粒体基因组结构图 Fig. 1 The mitochondrial genome structure of Scolopendra subspinipes dehaani ND1 -6/4l:NADH dehydrogenase subunit 1 -6/4l;ATP6/8:ATP synthase F0 subunit 6/8;COX1-3:cytochrome c oxidase subunit I-III;CYTB:cytochrome b;箭头方向表示基因的转录方向。 |
| 名称 | 起始位置 | 终止位置 | 长度(bp) | 链 | 反密码子 | 起始密码子 | 终止密码子 |
| tRNA-Ile | 1 | 70 | 70 | H | GAT | ||
| tRNA-Gln | 37 | 103 | 67 | L | TTG | ||
| tRNA-Met | 100 | 164 | 65 | H | CAT | ||
| ND2 CDS | 150 | 1100 | 951 | H | ATA | TAA | |
| tRNA-Trp | 1099 | 1158 | 60 | H | TCA | ||
| tRNA-Cys | 1133 | 1191 | 59 | L | GCA | ||
| tRNA-Tyr | 1180 | 1228 | 49 | L | GTA | ||
| COX1CDS | 1225 | 2755 | 1531 | H | ACG | T-- | |
| COX2 CDS | 2756 | 3436 | 681 | H | ATG | TAA | |
| tRNA-Lys | 3435 | 3491 | 57 | H | CTT | ||
| tRNA-Asp | 3486 | 3527 | 42 | H | GTC | ||
| ATP8 CDS | 3523 | 3675 | 153 | H | ATA | TAA | |
| ATP6 CDS | 3669 | 4332 | 664 | H | ATG | T-- | |
| COX3 CDS | 4333 | 5110 | 778 | H | ATG | T-- | |
| tRNA-Gly | 5111 | 5163 | 53 | H | TCC | ||
| ND3 CDS | 5164 | 5508 | 345 | H | ATA | TAG | |
| tRNA-Ala | 5507 | 5586 | 80 | H | TGC | ||
| tRNA-Arg | 5551 | 5616 | 66 | H | TCG | ||
| tRNA-Asn | 5587 | 5644 | 58 | H | GTT | ||
| tRNA-Ser | 5631 | 5682 | 52 | H | GCT | ||
| tRNA-Phe | 5717 | 5769 | 53 | L | GAA | ||
| ND5 CDS | 5771 | 7460 | 1690 | L | ATT | T-- | |
| tRNA-His | 7458 | 7506 | 49 | L | GTG | ||
| ND4 CDS | 7507 | 8812 | 1306 | L | ATT | T-- | |
| ND4L CDS | 8814 | 987 | 274 | L | ATA | T-- | |
| tRNA-Thr | 994 | 9168 | 75 | H | TGT | ||
| tRNA-Pro | 9121 | 9180 | 60 | L | TGG | ||
| ND6 CDS | 9203 | 9620 | 418 | H | ATA | T-- | |
| CYTB CDS | 9621 | 10707 | 187 | H | ATG | T-- | |
| tRNA-Ser | 10741 | 10798 | 58 | H | TGA | ||
| ND1 CDS | 10770 | 11708 | 939 | L | TTG | TAA | |
| tRNA-Leu | 11729 | 11798 | 70 | L | TAG | ||
| 16S rRNA | 11776 | 12995 | 1220 | L | |||
| 12S rRNA | 1342 | 13806 | 765 | L | |||
| tRNA-Val | 13199 | 13265 | 67 | L | TAC |
哈氏蜈蚣线粒体基因组全长14 538 bp,其中H链的碱基含量为:A=6 085 bp(41.9%),C=2 519 bp(17.3%),G=1 248 bp(8.6%),T=4 686 bp(32.2%),GC=3767 bp(25.9%),A>T>C>G,AT%>GC%。
2.4 蛋白编码基因哈氏蜈蚣线粒体基因组中蛋白编码基因全10 810 bp,约占全基因组的74.36%。A=4 542 bp(42.0%),C=1 924 bp(17.8%),G=958 bp(8.9%),T=3 386 bp(31.3%),GC=2 882 bp(26.7%),A>T>C>G,AT%>GC%。所有蛋白编码区均无内含子。除ND1以TTG为起始密码子,ND5和ND4以ATT为起始密码子,CO1以ACG为起始密码子, ND2、ATP8、ND3、ND4L、ND6以ATA起始外,其余均使用ATG为起始密码子。除ND3以TAG为终止密码子,ND2、COX2、ND1和ATP8以TAA为终止密码子,其余蛋白均以T碱基终止,并通过转录过程中3’端的poly A序列补全,形成完整的TAA终止密码子。氨基酸使用频率的分析见于表 2。在3 598个氨基酸中,氨基酸Cys(1.1%)、Gln(1.8%)和Arg(1.6%)出现频率较低,而频率最高的为Leu(14.0%)。
| Gene/AA | ND1 | ND2 | CO1 | CO2 | ATP8 | ATP6 | CO3 | ND3 | ND4L | ND4 | ND5 | ND6 | CYTB | 总计 |
| Ala | 11 | 10 | 38 | 9 | 0 | 7 | 13 | 3 | 1 | 13 | 20 | 3 | 17 | 145 |
| Cys | 6 | 1 | 0 | 2 | 0 | 0 | 1 | 1 | 2 | 13 | 12 | 1 | 2 | 41 |
| Asp | 6 | 1 | 16 | 10 | 0 | 1 | 3 | 4 | 3 | 8 | 18 | 0 | 6 | 76 |
| Glu | 12 | 2 | 7 | 10 | 0 | 4 | 8 | 4 | 3 | 9 | 6 | 2 | 5 | 72 |
| Phe | 33 | 18 | 40 | 12 | 3 | 14 | 24 | 13 | 12 | 52 | 68 | 9 | 34 | 332 |
| Gly | 21 | 10 | 41 | 6 | 0 | 6 | 15 | 3 | 9 | 38 | 43 | 4 | 20 | 216 |
| His | 0 | 5 | 16 | 6 | 0 | 5 | 13 | 0 | 1 | 7 | 5 | 1 | 10 | 69 |
| Ile | 33 | 33 | 51 | 23 | 6 | 21 | 22 | 12 | 6 | 40 | 44 | 19 | 42 | 352 |
| Lys | 7 | 14 | 8 | 3 | 4 | 8 | 5 | 8 | 1 | 7 | 6 | 5 | 8 | 84 |
| Leu | 46 | 53 | 57 | 27 | 1 | 36 | 29 | 22 | 14 | 75 | 69 | 27 | 48 | 504 |
| Met | 22 | 48 | 37 | 15 | 14 | 22 | 21 | 8 | 9 | 38 | 49 | 16 | 26 | 325 |
| Asn | 9 | 20 | 21 | 15 | 3 | 18 | 8 | 1 | 3 | 10 | 20 | 12 | 23 | 163 |
| Pro | 10 | 12 | 27 | 11 | 4 | 7 | 11 | 6 | 0 | 11 | 8 | 3 | 20 | 130 |
| Gln | 4 | 6 | 9 | 9 | 1 | 4 | 6 | 2 | 0 | 4 | 8 | 4 | 8 | 65 |
| Arg | 8 | 1 | 10 | 7 | 0 | 3 | 4 | 2 | 1 | 6 | 7 | 0 | 7 | 56 |
| Ser | 29 | 40 | 41 | 19 | 4 | 19 | 27 | 11 | 11 | 39 | 63 | 10 | 29 | 342 |
| Thr | 8 | 20 | 33 | 16 | 7 | 26 | 19 | 8 | 1 | 8 | 14 | 14 | 21 | 195 |
| Val | 20 | 3 | 27 | 10 | 0 | 10 | 10 | 3 | 8 | 22 | 40 | 0 | 16 | 169 |
| Trp | 8 | 8 | 14 | 6 | 2 | 4 | 12 | 2 | 2 | 11 | 21 | 2 | 10 | 102 |
| Tyr | 19 | 11 | 17 | 10 | 1 | 6 | 8 | 1 | 4 | 24 | 42 | 7 | 10 | 160 |
哈氏蜈蚣的12S rRNA和16S rRNA长度分别为765 bp和1 220 bp,GC含量分别为25.2%和24.9%。tRNA由MITOS[12]注释生成。20个tRNA基因中,最长的为tRNA-Ala(anticodon=TGC,80 bp),最短的为tRNA-Asp(anticodon=GTC,42 bp)。
2.6 非编码区哈氏蜈蚣线粒体基因组的控制区(control region)位于tRNA-Ile与12S rRNA之间,为732 bp。除控制区外,哈氏蜈蚣线粒体基因组中有6个非编码间隔区域,其中最长的间隔区长46 bp,位于12S rRNA和16S rRNA之间。
3 讨论本实验中采用差速离心以及滚环扩增的方法,得到纯度较高的线粒体DNA,打断后进行测序。用这种方法明显提升测序样品中线粒体DNA的比例,降低了细胞核基因的影响。相比于从总DNA测序结果中筛选拼接线粒体的方法[22-24],该方法对于线粒体DNA的测序深度显著增加,能够尽可能降低测序错误及假阳性结果的发生,得到更准确的线粒体基因组信息。
线粒体基因组具母系遗传特征,基因结构相对保守,其蛋白编码区及tRNA区结构变化在系统发育研究中应用较广[19]。本研究中仅发现20个tRNA, 与蜈蚣目已发表的其他物种相比[25],分别在tRNA-Ser与tRNA-Phe之间缺少一个tRNA-Glu、在ND1与tRNA-Leu之间缺少一个tRNA-Leu。tRNA的缺失可能是由于tRNA的截断或其二级结构不对称造成的[26]。这种现象的出现有多种可能的原因,如:缺少完全配对的受体茎、tRNA区与蛋白编码区或tRNA区的重叠、不对称的tRNA序列无法折叠成标准的三叶草结构等。在许多后生动物的线粒体基因组中都有类似现象出现[27-28]。
目前,蜈蚣目中仅有一个物种已发表线粒体基因组全序列,其近源物种的线粒体基因组研究未见报道。对哈氏蜈蚣线粒体基因组的研究,填补了蜈蚣科线粒体基因组数据的空白。未来更多近源物种数据的发表,将为药用蜈蚣的资源普查及保护提供可靠的依据。
| [1] | Undheim EA, King GF. On the venom system of centipedes (Chilopoda), a neglected group of venomous animals[J]. Toxicon, 2011, 57 (4): 512–524. DOI:10.1016/j.toxicon.2011.01.004 |
| [2] | Edgecombe G, Giribet G. Evolutionary biology of centipedes (Myriapoda:Chilopoda)[J]. Annual Review of Entomology, 2007, 52 (1): 151–170. DOI:10.1146/annurev.ento.52.110405.091326 |
| [3] | Uzel AP, Steinmann G, Bertino R, et al. Necrotizing fasciitis and cellulitis of the upper limb resulting from centipede bite:two case reports[J]. Chirurgie De La Main, 2009, 28 (5): 322–325. DOI:10.1016/j.main.2009.05.001 |
| [4] | Knysak I, Martins R, Bertim C R. Epidemiological aspects of centipede (Scolopendromorphae:Chilopoda) bites registered in greater S. Paulo, SP, Brazil[J]. Revista De Saúde Pública, 1998, 32 (6): 514. DOI:10.1590/S0034-89101998000600003 |
| [5] | Mohri S, Sugiyama A, Saito K, et al. Centipede bites in Japan[J]. Cutis, 1991, 47 (3): 189–190. |
| [6] | Fung HT, Lam SK, Wong OF. Centipede bite victims:a review of patients presenting to two emergency departments in Hong Kong[J]. Hong Kong medical journal, 2011, 17 (5): 381–385. |
| [7] | 方秀桐, 莫可元. 蜈蚣的药理研究进展[J]. 中国医药指南, 2015, 13 (18): 32–34. |
| [8] | 梁洁, 黄华营. 药用蜈蚣的化学成分及药理活性研究近况[J]. 广西中医药, 2005, 28 (4): 6–7. |
| [9] | Liu ZC, Zhang R, Zhao F, et al. Venomic and transcriptomic analysis of centipede Scolopendra subspinipes dehaani[J]. Journal of Proteome Research, 2012, 11 (12): 6197–6212. DOI:10.1021/pr300881d |
| [10] | 陈武忠, 曹海燕. 中药蜈蚣的研究进展[J]. 中国现代中药, 2011, 13 (7): 54–57. |
| [11] | 黄晓蒂, 陈园, 田莎, 等. 蜈蚣提取物对人肝癌细胞甲基化谱的影响[A]. 第八次全国中西医结合中青年学术论坛论文集[C]. 北京: 2016. |
| [12] | 姜璐璐, 曹艳华, 王诗敏, 等. 蜈蚣毒素的研究进展[J]. 西北药学杂志, 2009, 24 (6): 517–520. |
| [13] | 罗雷, 杨仕隆, 赖仞. 蜈蚣毒素研究进展[J]. 生命科学, 2016 (1): 27–32. |
| [14] | 周永芹, 韩莉. 中药蜈蚣的研究进展[J]. 中药材, 2008 (02): 315–319. DOI:10.3321/j.issn:1001-4454.2008.02.050 |
| [15] | 康四和, 邓海英, 江珍玉, 等. 我国药用蜈蚣分类鉴定及资源研究[J]. 中药材, 2016, 39 (4): 727–731. |
| [16] | 康四和, 邓海英, 刘义梅, 等. 我国药用蜈蚣资源与品质研究现状及对策[J]. 中南民族大学学报(自然科学版), 2016, 35 (2): 61–66. |
| [17] | 养小虫赚大钱本刊读者服务部举办中华土元、红龙蜈蚣、黄粉虫与竹鼠养殖技术培训班[J]. 农村百事通, 2013, 32(11): 84-85. |
| [18] | Liu ZC, Zhang R, Zhao F, et al. Venomic and transcriptomic analysis of centipede Scolopendra subspinipes dehaani[J]. Journal of Proteome Research, 2012, 11 (12): 6197–6212. DOI:10.1021/pr300881d |
| [19] | 左宝平. 少棘蜈蚣线粒体基因组全序列和平耳孔蜈蚣的地理种群遗传变异分析[D]. 南京: 南京师范大学, 2007. |
| [20] | 龚美蓉. 多足动物的系统发生关系及少棘蜈蚣线粒体基因组研究[D]. 南京: 南京师范大学, 2003. |
| [21] | 刘杰, 田晓轩, 崔英, 等. 药用动物乌梢蛇线粒体基因组全序列分析[J]. 天津中医药大学学报, 2016, 35 (3): 187–191. |
| [22] | Bernt M, Al E. MITOS:improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics & Evolution, 2013, 69 (2): 313–319. |
| [23] | Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9 (4): 357–359. DOI:10.1038/nmeth.1923 |
| [24] | Hahn C, Bachmann L, Chevreux B. Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads-a baiting and iterative mapping approach[J]. Nucleic Acids Research, 2013, 41 (13): e129. DOI:10.1093/nar/gkt371 |
| [25] | Gillett CPDT, Cramptonplatt A, Timmermans MJTN, et al. Bulk De Novo Mitogenome Assembly from Pooled Total DNA Elucidates the Phylogeny of Weevils (Coleoptera:Curculionoidea)[J]. Molecular Biology & Evolution, 2014, 31 (8): 2223–2237. |
| [26] | Tang M, Tan M, Meng G, et al. Multiplex sequencing of pooled mitochondrial genomes-a crucial step toward biodiversity analysis using mito-metagenomics[J]. Nucleic Acids Research, 2014, 42 (22): e166. DOI:10.1093/nar/gku917 |
| [27] | Gai Y, Ma H, Ma J, et al. The complete mitochondrial genome of Scolopocryptops sp. (Chilopoda:Scolopendromorpha:Scolopocryptopidae)[J]. Mitochondrial Dna, 2014, 25 (3): 192–193. DOI:10.3109/19401736.2013.792073 |
| [28] | Robertson HE, Lapraz F, Rhodes AC, et al. The Complete Mitochondrial Genome of the Geophilomorph Centipede Strigamia maritima[J]. Plos One, 2015, 10 (3): e0121369. DOI:10.1371/journal.pone.0121369 |
| [29] | Lavrov DV, Brown WM, Boore JL. A novel type of RNA editing occurs in the mitochondrial tRNAs of the centipede Lithobius forficatus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97 (25): 13738–13742. DOI:10.1073/pnas.250402997 |
| [30] | Masta SE, Boore JL. The complete mitochondrial genome sequence of the spider Habronattus oregonensis reveals rearranged and extremely truncated tRNAs[J]. Molecular Biology & Evolution, 2004, 21 (5): 893–902. |
2. Research and Development Center of Traditional Chinese Medicine, Tianjin International Joint Academy of Biomedicine, Tianjin 300457, China
 2018, Vol. 35
2018, Vol. 35