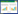文章信息
- 肖泽云, 李凯, 李爱秀
- XIAO Zeyun, LI Kai, LI Aixiu
- 中药化学数据库中HIV RT (NNRTI)/IN双靶点抑制剂的虚拟筛选
- Virtual screening of double target inhibitor of RT(NNRTI)/IN against HIV from traditional Chinese medicine database
- 天津中医药, 2018, 35(5): 386-391
- Tianjin Journal of Traditional Chinese Medicine, 2018, 35(5): 386-391
- http://dx.doi.org/10.11656/j.issn.1672-1519.2018.05.16
-
文章历史
- 收稿日期: 2018-02-12
2. 武警山西总队医院药剂科, 太原 030006;
3. 天津市职业与环境危害防制重点实验室, 天津 300309
艾滋病(AIDS)是由人类免疫缺陷病毒(HIV)引起的一种复杂疾病,其治疗一直是医学难题。HIV属于逆转录病毒,变异性强,在使用单靶点药物治疗AIDS的过程中,病毒容易产生耐药[1]。运用网络药理学对复杂疾病进行网络分析,发现AIDS等复杂疾病由多因素共同作用,其病理过程涉及多个细胞信号分子,仅针对单一细胞信号分子难以起到良好的治疗效果。多靶点药物能够多途径调节疾病的信号通路,同时作用于复杂疾病的多种细胞信号分子,提高治疗效果[2-3]。目前,临床上采用多药多靶点药物即“鸡尾酒”疗法(HAART)治疗AIDS。但多药联用毒副作用更大,容易引起不良反应,患者的依从性较差[1]。加之病毒交叉耐药的产生,影响了HAART的治疗效果,新型抗HIV药物的研发依然紧迫。与多药多靶点药物相比,单药多靶点药物为单一化学实体,药代动力学性质更好掌握,并且能够减少交叉耐药的发生。目前,虽然尚未有HIV单药多靶点抑制剂进入临床研究阶段,但单药多靶点药物已成为抗HIV创新药物的重要思路。
天然产物数量巨大,种类繁多,是研发新药和寻找先导化合物的重要来源。目前,在针对AIDS的天然产物研究中,发现了其中不乏对HIV病毒具有抑制作用的化合物。前期,总结了来源于天然产物的HIV单药多靶点抑制剂,这些抑制剂按照结构可以分为黄酮类、三萜及其苷类、苯丙素类、蛋白和肽类等,其中数量较多是黄酮类化合物,包括:蛇葡萄素(Ampelopsin)、灯盏花乙素(Scutellarin)、黄芩素(Baicalein)等[4]。这些抑制剂大多是针对HIV整合酶(IN)、逆转录酶(RT)和核糖核酸酶H(RNase H),以作用于IN链转移抑制剂(INSTI)作用位点和RT非核苷类逆转录酶抑制剂(NNRTI)结合部位的双靶点抑制剂[RT(NNRTI)/IN]居多。因此,天然产物对于HIV RT(NNRTI)/IN双靶点抑制剂的开发具有重要意义。
虚拟筛选[5]是指在计算机上,运用分子模拟技术和CADD方法,寻找与受体具有一定亲和力的小分子化合物,以提升筛选效率,缩短研发周期。选择3-OH HEPT类HIV RT(NNRTI)/IN双靶点抑制剂[6-7]展开研究,构建针对HIV RT(NNRTI)/IN双靶点抑制剂的分子相似性搜索模型,建立NNRTI和RT以及INSTI和IN对接模型[8-10],并基于两种筛选策略分别对TCMD[11]进行虚拟筛选:策略一,先后利用相似性搜索、利平斯基五规则和分子对接对TCMD逐步筛选,根据分子对接的打分高低排序;策略二,前期研究中基于所建的HIV RT(NNRTI)/IN双靶点抑制剂六点药效团模型和利平斯基五规则对TCMD进行筛选,得到数据库TCMD-SearchMD[10],再基于分子对接对TCMD-SearchMD进行筛选,根据打分高低排序。然后,将这两种策略筛选出的小分子化合物进行整理分析,以期从中发现潜在的HIV RT(NNRTI)/IN双靶点抑制剂。
1 材料 1.1 软件分子模拟与分子设计软件包MOE2009.10[12]和SYBYL8.1[13]。
1.2 数据库TCMD2003.1[11],包括9 127个化合物结构及其相关信息;本研究前期对TCMD筛选得到数据库TCMD-SearchMD[10],包括229个化合物结构及其相关信息。
1.3 受体结构从蛋白数据库(PDB)下载1JLA[14],为RT与HEPT类NNRTI TNK-651的复合物晶体结构,实验室基于IN与5ClTEP复合物晶体结构1QS4构建的理论模型1QS4(2Mg2+)[15]。
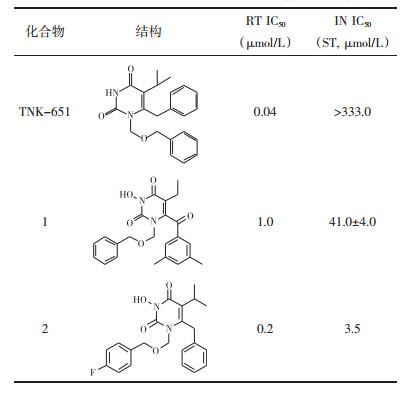
2 方法 2.1 分子相似性搜索模型的构建本研究分别选择TNK-651[16]和已知3-OH HEPT类HIV RT(NNRTI)/IN双靶点抑制剂[6-7]中活性较高的化合物1、2(见表 1)为模板分子,在MOE相似性搜索模块分别设定相似性系数(Tanimoto coefficient)为45%、50%和60%,对TCMD进行筛选,比较不同相似性系数命中的化合物,进而确定模板分子和对应的相似性系数。Molecular fingerprinting计算方法设定为MACCS,Force field为MMFF94x。
分别将配体分子TNK-651和5ClTEP从1JLA和1QS4(2Mg2+)中抽提出来,在SYBYL配体结构准备模块(ligand structure preparation)对配体分子进行加氢和能量优化。参数Force field选择Tripos,Method为Powell,Termination为Gardient 0.001 kcal/(mol·Å),Max Iterations为10 000。
2.2.2 受体准备受体准备在SYBYL Surflex-Dock蛋白结构准备模块(prepare protein structure)完成,首先将1JLA和1QS4(2Mg2+)配体分子TNK-651和5ClTEP抽提出来,保留A链,删除结晶水,对氨基酸残基进行加氢。然后,分别以1JLA和1QS4(2Mg2+)中配体的坐标为中心生成Protomol即结合口袋,保存结果用于后续研究。
2.2.3 分子对接为确定RT和NNRTI以及IN和INSTI最佳对接参数,在SYBYL Surflex-Dock模块,将配体分子TNK-651和5ClTEP分别对接回RT和IN活性位点。选择标准模式对接,设定TNK-651的结晶构象和5ClTEP结合构象为参比分子。调整对接参数设置,使配体分子对接构象最大程度重现复合物结构中配体分子原构象。一般以配体分子对接构象与复合物中结晶构象之间的均方根偏差(RMSD)值作为判断标准,当RMSD ≤ 2.0Å时,对接能够较好地重现出原配体分子的空间位置及构象。
2.3 数据库搜索TCMD库中包含9 127个化合物,为加快筛选速度,提高阳性率,制定两种筛选策略[17]。策略一:(1)运用构建的分子相似性搜索模型对TCMD进行筛选,设定化合物2为模板化合物,相似性系数为50%。(2)利用利平斯基五规则进行类药性分析。(3)将(2)筛选得到的化合物分别与RT和IN活性口袋对接。策略二:将TCMD-SearchMD中化合物分别与RT和IN活性口袋对接,对接采用筛选模式,参数设置与上文2.2一致。
3 结果 3.1 分子相似性搜索模板分子和相似性系数在构建分子相似性搜索模型的过程中,分别以TNK-651和化合物1、2为模板分子,选择相似性系数为60%、50%、45%对TCMD进行搜索,结果显示,比较发现当相似性系数为50%时,命中化合物的数量适中,用于下一步分子对接筛选。而化合物2活性较高,且对RT和IN抑制活性更为均衡。综合考虑,本研究选择以化合物2为模板分子,相似性系数为50%的分子相似性搜索结果用于后续研究。见表 2。
本研究分别构建NNRTI和RT活性位点以及INSTI和IN活性位点对接模型,通过调整对接参数,使配体和酶达到最合理的相互作用。结果显示,TNK-651与RT对接得分为10.60,对接构象与1JLA中结晶构象空间距离的RMSD值为0.977Å。5ClTEP与IN对接得分为3.98,对接构象与1QS4(2Mg2+)中原构象空间距离的RMSD值为1.187Å。可以看出,空间距离的RMSD值均在2Å以下,提示对接构象比较完整地重现了配体分子原构象的空间位置,程序适用,参数设置合理。对接参数设置如下:Max conformations per Fragment为20,Minimum RMSD Between Final Poses为0.05Å,Maximum Number of Poses per Ligand为20。
3.3 筛选结果 3.3.1 基于策略一的筛选结果本研究将相似性搜索命中的152个化合物利用利平斯基五规则筛选,命中100个小分子。100个小分子中51个与IN对接打分大于4,有43个与RT对接打分大于4,与IN和RT对接打分均大于4的有28个。其中,与IN对接打分大于6的有8个,与RT对接打分大于6的有14个,与IN和RT对接打分均在6以上的有7个。见表 3。
|
将TCMD-SearchMD中小分子分别与IN和RT进行对接,与IN对接打分在4以上的有158个,与RT对接打分在4以上的有195个,与IN和RT对接打分均大于4的有143个;其中,与IN对接打分在6以上的有47个,与RT对接打分在6以上的有121个,与IN和RT对接打分均大于6的小分子有29个。见表 4。

|
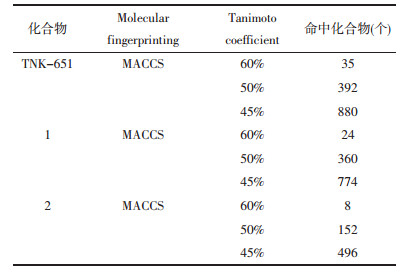
综合筛选结果,除去1个相同的生物碱类化合物M7351,得到35个潜在HIV RT(NNRTI)/IN双靶点抑制剂,与RT和IN对接得分均大于6,对抑制剂的结构类型进行总结,包括黄酮类(15个)、生物碱类(8个)、多酚类(3个)、苯丙素类(3个)、苷类(3个)、有机酸类(2个)和醌类(1个),黄酮类化合物占到了42.86%,包括蛇葡萄素、麝香草素、车前子苷等,而策略一对TCMD筛选命中的7个化合物全部为生物碱类。与RT和IN对接结合较好的化合物蛇葡萄素结构如图 1(a)所示,图 1(b,c)为其与RT活性位点相互作用2D、3D示意图,可以看出除疏水相互作用外,蛇葡萄素糖环3-OH与Lys101、Lys103之间形成氢键相互作用,糖环1-位苯环与Tyr188之间形成π-π相互作用。观察蛇葡萄素与IN活性位点相互作用[见图 1(d、e)],可以看出其与IN之间除存在疏水相互作用和氢键相互作用外,还通过氨基酸残基Asp64和Asp116共同与双Mg2+发生金属离子螯合作用。
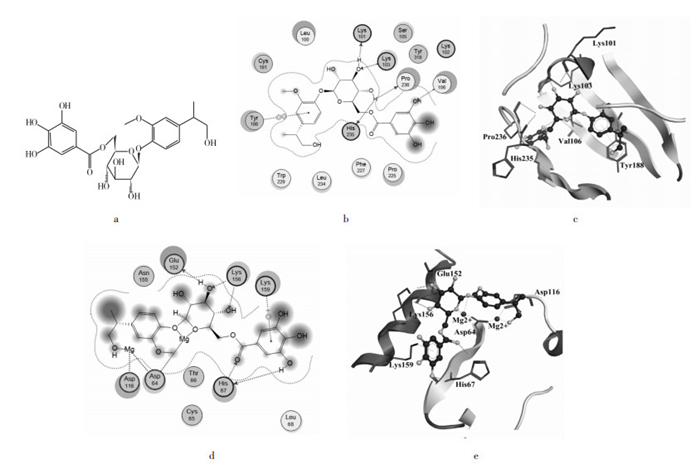
|
| 图 1 蛇葡萄素结构式(a);与RT相互作用2D图(b);与RT相互作用3D图(c);与IN相互作用2D图(d);与IN相互作用3D图(e) Fig. 1 Structure of Ampelopsin (a); 2D diagram of the interaction of Ampelopsin with RT (b); 3D diagram of the interaction of Ampelopsin with RT (c); 2D diagram of the interaction of Ampelopsin with IN (d); 3D diagram of the interaction of Ampelopsin with IN (e) |
不同的筛选方法具有各自的针对性和优缺点,每种方法均有可能漏筛。本研究通过两种策略对TCMD进行筛选,每种策略既可以筛选到相同的化合物,也可以筛选到不同的化合物。采用两种策略,能够相互补充,避免漏筛。TCMD库包含9 127个化合物,本研究首先采用相似性搜索和药效团筛选,接着运用利平斯基五规则分析,得到的小分子分别与RT和IN进行对接,最终保留潜在的HIV RT(NNRTI)/IN双靶点抑制剂。已有研究表明,利用Surflex-Dock程序进行分子对接,打分>4时配体与受体之间存在较强的相互作用,打分>6则可以预测配体对受体的抑制活性达微摩尔级。为加快新药发现的速度和减少不必要的浪费,本研究优先选择与RT和IN对接打分均大于6的化合物进行分析,以确定率先展开实验研究的化合物。从数量上看,命中的黄酮类化合物占到了42.86%,是开发潜在HIV RT(NNRTI)/IN双靶点抑制剂的主要来源,与文献报道天然产物中HIV RT(NNRTI)/IN双靶点抑制剂中黄酮类化合物居多一致。
本研究的筛选结果与其他研究者实验研究也有契合点,如实验表明杜仲提取物能够抑制HIV gp41六螺旋结构的形成,是潜在的HIV抑制剂[18],本研究筛选得到来源于杜仲的酚类化合物Koaburaside,Koaburaside具有潜在的HIV RT和IN抑制活性,可能是杜仲提取物抗HIV的物质基础。苦参中的黄酮类化合物苦参新醇H(Kushenol H)具有弱抗单纯性疱疹病毒HSV-1和HSV-2活性[19],本研究筛选结果显示其与HIV RT和IN对接打分均大于6,预测对RT和IN抑制活性在微摩尔级。此外,化合物蛇葡萄素能抑制HIV的感染,还能干扰HIV对CD4+细胞的黏附[20],本研究对接结果表明蛇葡萄素与RT和IN结合较好,并与活性位点之间形成了关键相互作用,表明其可能是通过作用于HIV RT和IN,发挥抗HIV活性。
综上,本研究运用分子模拟技术和CADD方法构建了针对TCMD的虚拟筛选模型和策略,检出35个潜在的HIV RT(NNRTI)/IN双靶点抑制剂,为天然产物来源的HIV双靶点药物研发提供了理论基础和发展方向。
| [1] | Obermeier M, Ehret R, Wienbreyer A, et al. Resistance remains a problem in treatment failure[J]. Journal of the International AIDS Society, 2014, 17 (4): 161–162. |
| [2] | 李爱秀. 多靶点药物的研究及筛选策略的提出[J]. 武警后勤学院学报:医学版, 2013, 22 (11): 1030–1034. |
| [3] | 展鹏, 刘新泳, 李潇, 等. 抗艾滋病药物设计新策略:多靶点及多价态结合配体[J]. 中国药物化学杂志, 2013, 23 (5): 406–416. |
| [4] | 李凯, 李爱秀, 靳玉瑞, 等. 天然产物来源的抗HIV-1多靶点抑制剂研究进展[J]. 中草药, 2015, 46 (12): 1840–1848. DOI:10.7501/j.issn.0253-2670.2015.12.025 |
| [5] | 叶德泳. 药物设计学[M]. 北京: 高等教育出版社, 2015: 8. |
| [6] | Tang J, Maddali K, Dreis CD, et al. 6-Benzoyl-3-hydroxypyrimidine-2, 4-diones as Dual Inhibitors of HIV Reverse Transcriptase and Integrase[J]. Bioorganic & Medicinal Chemistry Letters, 2011, 21 (8): 2400–2402. |
| [7] | Tang J, Maddali K, Dreis CD, et al. N-3 nydroxylation of pyrimidine-2, 4-diones yields dual inhibitors of HIV reverse transcriptase and integrase[J]. Acs Medicinal Chemistry Letters, 2011, 2 (1): 63–67. DOI:10.1021/ml1002162 |
| [8] | Islam MA, Pillay TS. Exploration of the structural requirements of HIV-protease inhibitors using pharmacophore, virtual screening and molecular docking approaches for lead identification[J]. Journal of Molecular Graphics &Modelling, 2015, 56 (2): 20–30. |
| [9] | Wu KZ, Li AX, Liu XT, et al. Building the pharmacophore model of HIV-1 integrase strand transfer inhibitors and studying their inhibition mechanism[J]. Chinese Journal of Chemistry, 2010, 29 (4): 575–581. |
| [10] | 肖泽云, 李凯, 李爱秀, 等. 新型HIV逆转录酶和整合酶双靶点抑制剂的虚拟筛选[J]. 中国医药导报, 2017, 14 (27): 4–7. |
| [11] | 周家驹, 谢桂荣, 严新建. 中药原植物化学成分手册[M]. 北京: 化学工业出版社, 2004: 9. |
| [12] | 朱瑞新. 计算机辅助药物设计:基本方法原理概要与实践详解[M]. 大连: 大连理工大学出版社, 2011. |
| [13] | Belekar V, Shah A, Garg P. High-throughput virtual screening of phloroglucinol derivatives against HIV-reverse transcriptase[J]. Molecular Diversity, 2013, 17 (1): 97–110. DOI:10.1007/s11030-012-9417-7 |
| [14] | Ren J, Nichols C, Bird L, et al. Structural mechanisms of drug resistance for mutations at codons 181 and 188 in HIV-1 reverse transcriptase and the improved resilience of second generation non-nucleoside inhibitors[J]. Journal of Molecular Biology, 2001, 312 (4): 795–805. DOI:10.1006/jmbi.2001.4988 |
| [15] | 吴可柱, 李爱秀, 缪有盼, 等. HIV-1整合酶四聚体结构模拟及其活性位点分析[J]. 中国生物化学与分子生物学报, 2009, 25 (6): 549–555. |
| [16] | 孟歌, 陈芬儿. HEPT类HIV-1逆转录酶抑制剂的研究进展[J]. 中国药物化学杂志, 2004, 14 (1): 56–64. |
| [17] | Distinto S, Esposito F, Kirchmair J, et al. Identification of HIV-1 reverse transcriptase dual inhibitors by a combined shape-, 2D-fingerprint-and pharmacophore-based virtual screening approach[J]. European Journal of Medicinal Chemistry, 2012, 50 (50): 216–229. |
| [18] | 吕琳, 孙燕荣, 徐伟, 等. 杜仲提取物抗HIV活性成分的分离鉴定[J]. 中药材, 2008, 31 (6): 847–850. |
| [19] | Woo ER, Hwak JH, Kim HJ, et al. A new prenylated flavonol from the roots of Sophora flavescens[J]. Journal of Natural Products, 1998, 61 (12): 1552–1554. DOI:10.1021/np980103j |
| [20] | Liu DY, Ye JT, Yang WH, et al. Ampelopsin, a small molecule inhibitor of HIV-1 infection targeting HIV entry[J]. Biomedical and Environmental Sciences, 2004, 17 (2): 153–164. |
2. Pharmacy Department, Shanxi General Hospital of PAP, Taiyuan 030006, China;
3. Tianjin Key Laboratory for Prevention and Control of Occupational and Environmental Hazard, Tianjin 300309, China
 2018, Vol. 35
2018, Vol. 35