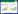文章信息
- 张昆林, 孙鹏程, 陈雪梅, 陈建新, 张妍, 王一, 王济, 王琦
- ZHANG Kunlin, SUN Pengcheng, CHEN Xuemei, CHEN Jianxin, ZHANG Yan, WANG Yi, WANG Ji, WANG Qi
- 基于生物信息学方法的祛风解表中药干预过敏体质机制研究
- Research on the mechanism of intervention of allergic constitution (TIZHI) by Chinese medicines for expelling wind to relieve superficies based on bioinformatics
- 天津中医药, 2021, 38(3): 273-279
- Tianjin Journal of Traditional Chinese Medicine, 2021, 38(3): 273-279
- http://dx.doi.org/10.11656/j.issn.1672-1519.2021.03.01
-
文章历史
- 收稿日期: 2020-12-11
2. 中国科学院大学心理学系, 北京 100049;
3. 北京中医药大学中医学院, 北京 100029;
4. 北京中医药大学国家中医体质与治未病研究院, 北京 100029
过敏性疾病又称变态反应性疾病,在过去几十年中发病率显著上升,世界变态反应组织(WAO)2004年在全球33个国家(未含中国、印度等)的流行病学调查结果[1]显示:在这些国家的12亿总人口中,22%患有特异性免疫球蛋白E(IgE)介导的过敏性疾病,如过敏性鼻炎、哮喘、结膜炎、湿疹等,影响儿童和成人的生活质量,严重全身过敏反应甚至危及生命。WAO在2013年发布的《WAO White Book on Allergy》[2]中指出,在发达国家和发展中国家,全世界变态反应性疾病的流行率都在急剧上升,应将过敏作为一大公共卫生问题,并在非传染性疾病的框架内加以解决。
目前西医学主要使用脱敏及对症治疗的方法控制过敏,常用药物包括减充血剂、皮质激素类、肥大细胞稳定剂、抗胆碱药、抗组胺药以及白三烯受体拮抗剂等,这些药物作用效果快,但作用位点单一,常见不良反应有头晕、嗜睡、口干恶心等,且长期使用容易产生耐药性等弊端[3-4]。对于频发、多发的过敏性疾病,现有研究多从病、证、病机的角度阐发中医药治疗策略。王琦院士提出“辨体-辨病-辨证相结合”的诊疗模式,构建过敏体质理论,指出过敏体质是机体易发过敏性疾病的根本原因,防治过敏性疾病的核心在于调节“体质土壤”[5-6]。近年来西方学者的研究印证了过敏体质的存在。Goks ö r等[7]指出,过敏的进程包括过敏性疾病相关模式的共存,而不仅仅是单一过敏性疾病的逐步发展。Ferreira等[8]的研究表明,湿疹、花粉症和哮喘有共同的遗传风险基因。因此过敏体质是过敏进程的内在原因,须改变治疗“过敏病”的观念,确立治疗“过敏人”的思想,以干预过敏体质来防治过敏性疾病,是中医药从根本上解决过敏的好方法[5]。
从实验研究的角度,中药的抗过敏作用已得到广泛验证。研究表明天然药物中包含的多种黄酮类化合物、萜类化合物、醌类化合物等有良好的抗过敏活性[4]。目前临床使用较多的为祛风解表等药物,归肺经的解表药居于首位[9]。研究发现祛风中药能在抑制IgE产生、保护和稳定靶细胞膜、对抗过敏介质、中和变应原等多个环节起作用[10]。因此本研究以“祛风解表”作为研究视角,将体质与药物相结合,运用网络药理学、多组学联合通路分析等方法构建药物-靶点-通路网络,以期揭示中药调控过敏体质的分子机制。
1 材料与方法 1.1 解表祛风中药提取药物靶点在中国知网(CNKI)以“抗过敏中药”为关键词检索,筛选有实验研究支撑的单味中药。通过TCMSP数据库[11],以口服生物利用度OB≥20%,类药性(DL)≥0.10为标准筛选活性成分。使用PharmMapper数据库[12]进行靶点预测。以Norm.Fit > 0.8 & zscore > 0.5为阈值筛选靶点,使用UniProt数据库[13]将筛选后的靶点转化为Entrez ID及Gene Symbol。
1.2 GWAS catalog提取过敏易感基因下载GWAScatalog数据(版本号:e100_r2020-12-02),从中提取与3种典型过敏性疾病(哮喘、过敏性鼻炎、湿疹)都相关的易感基因(P < 5e-8)[14]。并加入Ferreira等[8]通过GWAS结果预测的这3种过敏性疾病的功能性基因。
1.3 过敏体质表达芯片数据提取差异基因基于已有数据,从过敏体质基因芯片表达数据[15]中提取差异基因。该数据来自5个过敏体质(过敏性鼻炎)病例和5个健康对照。取外周血分离树突细胞,体外培养诱导成熟,对两组成熟前后的树突细胞分别用Agilent Human lncRNA(4*180K)芯片检测,总计20个样本数据。鉴别出成熟前(imDEG)、成熟后(mDEG)两组差异基因。差异基因为表达差异 > 1.5倍及线性模型P值< 0.05的基因。
1.4 多组学联合通路分析使用KOBAS 3.0[16]对以上4个基因集(靶蛋白/GWAS易感基因/imDEG/mDEG)分别做通路富集分析,采用超几何分布检验,通路数据库取KEGG、REACTOM、BioCyc、PANTHER、GO,共检测6 268个通路。使用Python 2.7进行多组学联合分析,参照PaintOmics[17]多组学联合通路分析的思想对每一个通路的4个统计检验结果进行P值合并(元分析)。因为数据涉及同一组学数据内部平权和不同组学数据之间的平权,所以采用Stouffer’s Z-score方法[18-19]进行P值合并,基于该方法对P值进行整合的元分析方法已经收录于常用统计遗传学工具并被广泛使用[20-23]。取靶蛋白、GWAS基因、imDEG、mDEG通路的权重分别为1/3、1/3、1/6、1/6。最后用Bonferroni校正法(P值乘以检验次数)对元分析的P值进行校正。取在4个基因集中均包含2个及以上基因、总基因数不超过200[24]且校正P值< 0.05的通路作为多组学显著性通路。
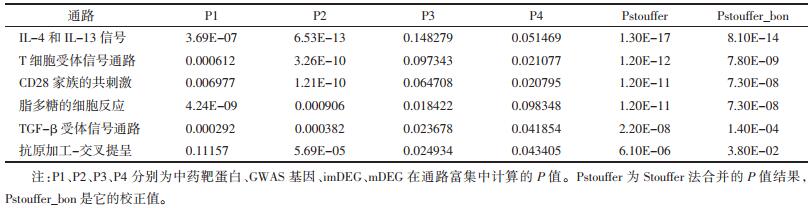
1.5 核心靶点筛选及药物-靶点-通路网络构建将过敏相关多组学显著通路涉及的药物靶点导入STRING(Version:11.0)[25]数据库,建立蛋白-蛋白相互作用网络(PPI)。导入Cytoscape 3.8.0[26],使用cytoHubba插件[27]计算MCC(Maximal Clique Centrality,最大集团中心度)算法得分。将结果与Mcode插件[28]筛选的子网络(默认参数)相印证,确定核心靶点(hub gene)。
2 结果 2.1 药物活性成分及靶点筛选结果筛选到15味有抗过敏作用的解表祛风药,发散风寒药包括麻黄、桂枝、紫苏叶、生姜、荆芥、防风、白芷、细辛、苍耳子、辛夷、鹅不食草,发散风热药包括薄荷、蝉蜕、桑叶、葛根。共筛选出545个活性成分,预测获得1 602个靶点,按阈值筛选后,剔除未匹配到UniProt ID的结果并去重后获得225个靶点。转化为Entrez ID及Gene Symbol后共计226个基因。
2.2 GWAS catalog提取基因结果从GWAS catalog数据中提取到易感基因356个。加入Ferreira等[8]预测的132个功能基因,合并去重后得到427个基因。
2.3 过敏体质表达芯片数据提取基因结果树突状细胞成熟前后的病例组和健康组分别鉴定出1 240个(imDEG)和743个(mDEG)差异基因。
2.4 多组学通路分析结果靶蛋白/GWAS基因/imDEG/mDEG的单组学通路分析分别得到3 860、3 406、2 688、1 563个通路,合并去重后共6 268个通路,交集为582个通路。在4个基因集中均包含两个及以上基因、总基因数不超过200且校正P值Pstouffer_bon < 0.05的多组学显著性通路共有24个。结合文献,确定6个过敏相关显著性通路,对应药物的28个靶点蛋白。通路为:白细胞介素(IL)-4和IL-13信号(R-HSA-6785807)、T细胞受体信号通路(GO:0050852)、CD28家族的共刺激(R-HSA-388841)、脂多糖的细胞反应(GO:0071222)、转化生长因子(TGF)-β受体信号通路(GO:0007179)、抗原加工-交叉提呈(R-HSA-1236975)。见表 1。
28个靶点蛋白中确定11个核心靶点。见表 2。
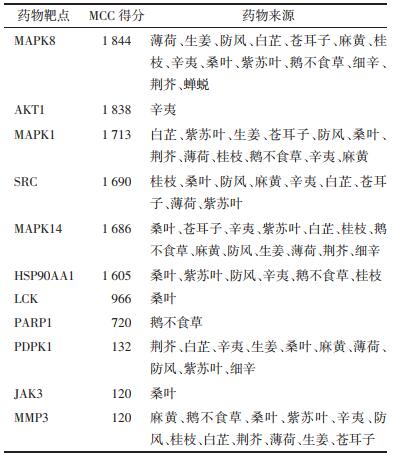
将15味药物、28个靶点、6条通路的数据导入Cytoscape,构建药物-靶点-通路网络。见图 1。
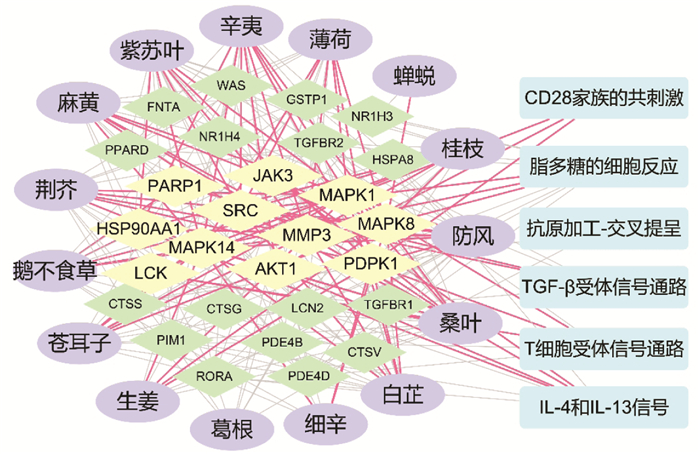
|
| 椭圆形代表 15味药物,菱形代表 28个靶点(黄色代表 11个核心靶点),长方形代表 6条过敏通路。 图 1 药物-靶点-通路关系图 Fig. 1 Drug-target-pathway interaction |
过敏反应中,首次接触具有过敏原的个体黏膜部位后,便建立了敏化阶段。通过驻留在各个黏膜组织中的树突状细胞摄取和呈递过敏原,可以激活幼稚的CD4+ T细胞并分化为2型辅助T细胞(Th2)[29]。交叉提呈是免疫系统监测组织和吞噬细胞是否存在异物抗原的主要机制。抗原提呈细胞,如树突状细胞,是赋予交叉提呈抗原能力的主要细胞[30]。在1型过敏反应下,树突状细胞的提呈导致Th2细胞驱动的针对过敏原的特异性免疫应答[29]。
主要组织相溶性复合体(MHC)分子对关联抗原肽的特异性识别会触发T细胞受体(TCR)信号转导,T细胞上的共刺激受体和共抑制受体确定T细胞功能并决定TCR信号转导的功能结果[31]。TCR激活可促进许多信号转导级联反应,许多TCR信号通路的破坏可能导致Th2表型,进而驱动机体过敏性疾病[32]。
T淋巴细胞的激活被认为至少需要两个信号,一个信号由抗原识别后T细胞受体复合物传递,另一个由共刺激受体(如CD28)参与时提供[33]。共刺激信号通路主要影响T细胞的活化、增殖和分化,并导致Thl/Th2分化比例失衡,在过敏性疾病的发病过程中起着重要作用[34]。
IL-4和IL-13是由Th2细胞、肥大细胞、嗜酸性细胞产生的2种细胞因子,它们与哮喘和过敏密切相关。由IL-4和IL-13驱动的Th2细胞及其细胞因子的产生促进了B细胞过敏原IgE抗体的产生[29]。IL-4和IL-13还能激活JAK/STAT信号级联反应,对过敏反应有促进作用[35]。
TGF-β是生长因子超家族中具有免疫负调控功能的一大类细胞因子家族,在调节炎症反应、伤口愈合、免疫稳态及免疫耐受中发挥重要作用[36]。过敏性疾病发展的关键机制涉及对调节性T(Treg)细胞介导的免疫耐受的干扰,Turner等[37]最新的一项研究证明了Treg细胞来源的TGF-β1在调节变态反应和自身免疫反应中的关键作用。
脂多糖(LPS)作为革兰阳性菌细胞壁成分,在人类工作或生活环境中几乎无处不在,是一种强烈的免疫调节物质,能促进Th1型反应而抑制Th2型反应,与过敏性哮喘呈负相关。同时有研究表明LPS具有对Th1/Th2平衡有双向调节作用[38]。
3.2 祛风解表药物核心靶点分析丝裂原活化蛋白激酶(MAPK8)、MAPK1、MAPK14均是MAPK家族的成员。MAPK是生物体内重要的信号转导系统之一,能被多种炎性刺激所激活,并对炎症的发生、发展起重要调控作用[39]。在T细胞中,MAPK8和MAPK9是Th细胞分化为Th1细胞所必需的。动物实验发现MAPK8基因敲除小鼠缓解了特应性皮炎样症状[40]。MAPK1/ERK2和MAPK3/ERK1是在MAPK/ERK级联中起重要作用的2个MAPK。ERK1/2与包括Th2分化和增殖在内的过敏性炎症有关。气道中ERK1/2 MAPK的磷酸化引起炎症和结构变化,从而导致T细胞活化、嗜酸性粒细胞和肥大细胞浸润、支气管高反应性和气道重塑[41]。MAPK14/p38α调节多个T细胞受体相关信号,并对Th2分化和过敏性炎症产生负面影响[42]。
基质金属蛋白酶(MMP)是由Ca2+激活的、依赖Zn2+的内肽酶家族,具有降解细胞外基质(ECM)和基底膜各种成分的能力。它们与正常的生理组织重塑、炎症和肿瘤扩散有关。MMP-1、MMP-2、MMP-3、MMP9与肠黏膜炎症相关[43]。MMP-3和MMP-9与哮喘的慢性气道改变有关,可以通过释放和激活潜在的基质结合生长因子来增强气道纤维化和平滑肌增殖[44]。
蛋白激酶B(AKT1)是称为AKT激酶的3种紧密相关的丝氨酸/苏氨酸蛋白激酶(AKT1,AKT2和AKT3)之一。AKT1参与哮喘气道平滑肌的重塑。质粒转导或慢病毒感染引起的AKT1过表达会导致培养的犬气道心肌细胞的大小和蛋白质含量逐渐增加,表明AKT1的激活可能导致哮喘的气道平滑肌肥大。AKT1可能是预防或逆转哮喘气道平滑肌肥大的有用治疗靶标[45]。
PDPK1通过磷酸化作用调节一组相关的蛋白激酶的活性,包括PKB/Akt、S6K、RSK、SGK以及PKC等。其中PKC是过敏原及肿瘤坏死因子(TNF)-α等促炎因子的重要靶标,PKC活化后可引起促炎信号的累积,如NF-κB的激活,中药成分如牛蒡子苷元等可以通过抑制PDPK1阻断MAPK通路的信号转导,抑制NF-κB基因的表达[46]。PDK1可以减轻豚鼠气管分离肌的收缩,作为哮喘靶标可能有益于气流阻塞[47]。PDK1可能作为嗜酸性粒细胞炎症的新型治疗靶标[48]。
Src家族激酶是非受体蛋白酪氨酸激酶。Src激酶与哮喘发病机制中涉及的细胞活化有关,参与肥大细胞、嗜酸性粒细胞和其他细胞的激活。Src在TCR刺激后的信号通路激活和转导中起重要作用,在T细胞发育、分化、增殖和存活中至关重要[49]。Lck是Src家族成员。鉴于Lck在细胞因子产生和T细胞信号转导中起主要作用,Lck表达和活性的改变可能导致各种疾病,例如哮喘、牛皮癣、炎症性肠病等,可以确立为各种炎症介导的病理生理条件下的治疗靶标之一[50]。
Hsp90是真核生物中特征明确,保守且必不可少的伴侣蛋白。Hsp90α是Hsp90的亚型,Hsp90α除具有细胞内作用外,目前还被认为在伤口愈合和炎症中起分泌性细胞外作用[51]。由于其结合与解离与炎症因子信号通路中许多信号分子的非活性/活性结构密切相关,可以作为炎症信号通路的研究靶分子[52]。
JAK3是非受体酪氨酸激酶JAK家族成员,在细胞的生长、存活、发育、分化、增殖、凋亡及免疫调节等方面起重要作用。JAK3对树突状细胞的成熟和耐受性起着至关重要的作用[53]。在特应性皮炎中,JAK1、JAK2和JAK3明显过表达。JAK/STAT途径在多种炎症性皮肤病中具有重要作用,JAK3被认为炎症性皮肤病的潜在靶点[54]。
PARP是真核细胞内具有多聚腺苷酸二磷酸核糖基(PAR)催化活性的蛋白酶,与炎症、肿瘤、衰老等疾病相关联。在树突细胞内,PARP1破坏树突细胞免疫活性[55]。PARP与人的哮喘和过敏性鼻炎有遗传相关性,抑制PARP可能成为抗过敏性哮喘的未来治疗策略[56]。
4 结论本研究从过敏体质入手,综合运用多种生物信息学分析方法,首次在通路水平将来自3个不同数据源(中药的预测靶点、GWAS提取的过敏共病基因、基因芯片鉴定的过敏体质差异基因)的基因结果集进行相互印证,结合基因组学和转录组学数据信息,综合检测过敏过程中靶蛋白可能参与的通路。通过元分析合并P值并加权校正,筛选出6条多组学显著性通路。这些通路主要反映了树突状细胞的提呈、T细胞受体信号转导、Th2型细胞因子分泌以及免疫耐受的过敏进程。通过将这6条通路涉及的28个靶点建立蛋白互作网络,建立药物-靶点-通路网络,筛选出11个核心靶点,它们基本是免疫、炎症、过敏的相关重要分子,是祛风解表中药调控过敏体质的可能关键节点。
本研究突破了中药调控单一过敏性疾病的研究模式,将“祛风解表”的临床治法和“体质土壤”的疾病机制相关联,鉴定了祛风解表中药干预过敏体质的关键通路和关键靶点,有助于中医药治疗过敏机制研究的方法学革新,推进疾病医学向体质医学的发展与转化,以期发挥中医药特色与优势应对全球过敏危机。同时研究也存在药物和基因集数据量偏少、缺少实验验证等不足,期待后续研究中进行完善。
| [1] |
WARNER J O, KALINER M A, CRISCI C D, et al. Allergy practice worldwide: a report by the World Allergy Organization Specialty and Training Council[J]. International Archives of Allergy and Immunology, 2006, 139(2): 166-174. DOI:10.1159/000090502 |
| [2] |
PAWANKAR R, CANONICA G, HOLGATE S, et al. World allergy organization (WAO) white book on allergy, update[M]. 2013.
|
| [3] |
韦森春. 过敏性鼻炎的治疗药物研究进展[J]. 中国医药指南, 2013, 11(20): 497-498. WEI S C. Advances in treatment of allergic rhinitis[J]. Guide of China Medicine, 2013, 11(20): 497-498. |
| [4] |
连晨蕾, 丛慧, 吴雅清, 等. 抗过敏天然产物研究进展[J]. 中国药学杂志, 2017, 52(12): 997-1004. LIAN C L, CONG H, WU Y Q, et al. Research progress of anti-allergy natural products[J]. Chinese Pharmaceutical Journal, 2017, 52(12): 997-1004. |
| [5] |
王琦, 骆庆峰. 过敏体质的概念、形成与调控原理[J]. 北京中医药大学学报, 2004, 27(2): 6-8. WANG Q, LUO Q F. Concept, formation and regulation of allergic constitution[J]. Journal of Beijing University of Traditional Chinese Medicine, 2004, 27(2): 6-8. |
| [6] |
李玲孺, 张惠敏, 王济, 等. 王琦辨体-辨病-辨证治疗过敏性疾病经验[J]. 中医杂志, 2012, 53(20): 1720-1723. LI L R, ZHANG H M, WANG J, et al. WANG Qi's experience in treating allergic diseases based on constitution-disease-syndrome differentiation[J]. Journal of Traditional Chinese Medicine, 2012, 53(20): 1720-1723. |
| [7] |
GOKSÖR E, LOID P, ALM B, et al. The allergic march comprises the coexistence of related patterns of allergic disease not just the progressive development of one disease[J]. Acta Paediatrica, 2016, 105(12): 1472-1479. DOI:10.1111/apa.13515 |
| [8] |
FERREIRA M A, VONK J M, BAURECHT H, et al. Shared genetic origin of asthma, hay fever and eczema elucidates allergic disease biology[J]. Nature Genetics, 2017, 49(12): 1752-1757. DOI:10.1038/ng.3985 |
| [9] |
李彦军, 赵学萍, 肖遥, 等. 从祛风药论风邪在过敏性哮喘发病中的重要作用[J]. 吉林中医药, 2012, 32(7): 721-723. LI Y J, ZHAO X P, XIAO Y, et al. The important role of wind pathogen in the pathogenesis of allergic asthma from the perspective of anti-wind drugs[J]. Jilin Journal of Traditional Chinese Medicine, 2012, 32(7): 721-723. |
| [10] |
邓德厚, 沈小珩. 祛风中药治疗过敏性疾病实验研究进展[J]. 中国中医药信息杂志, 2007, 14(8): 92-94. DENG D H, SHEN X H. Progress in experimental research on the treatment of allergic diseases with Chinese medicine for dispelling wind[J]. Chinese Journal of Information on Traditional Chinese Medicine, 2007, 14(8): 92-94. |
| [11] |
RU J, LI P, WANG J, et al. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines[J]. Journal of Cheminformatics, 2014, 6(1): 13. DOI:10.1186/1758-2946-6-13 |
| [12] |
WANG X, SHEN Y, WANG S, et al. PharmMapper 2017 update: a web server for potential drug target identification with a comprehensive target pharmacophore database[J]. Nucleic Acids Research, 2017, 45(W1): W356-W360. DOI:10.1093/nar/gkx374 |
| [13] |
THE UNIPROT CONSORTIUM. UniProt: a worldwide hub of protein knowledge[J]. Nucleic Acids Research, 2019, 47(D1): D506-D515. DOI:10.1093/nar/gky1049 |
| [14] |
BUNIELLO A, MACARTHUR J A L, CEREZO M, et al. The NHGRI-EBI GWAS Catalog of published genome-wide association studies, targeted arrays and summary statistics 2019[J]. Nucleic Acids Research, 2019, 47(D1): D1005-D1012. DOI:10.1093/nar/gky1120 |
| [15] |
陈雪梅. 中医过敏体质树突状细胞成熟lncRNA-mRNA表达及蛋白质芯片研究[D]. 北京: 北京中医药大学, 2015. CHEN X M. Research on immature and mature dendritic cells of TCM allergy constitution (TIZHI) based on lncRNA-mRNA chip and protein chip[D]. Beijing: Beijing University of Chinese Medicine, 2015. |
| [16] |
XIE C, MAO X, HUANG J, et al. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 2011, 39(Suppl 2): W316-322. |
| [17] |
HERNÁNDEZ-DE-DIEGO R, TARAZONA S, MARTÍNEZ-MIRA C, et al. PaintOmics 3:a web resource for the pathway analysis and visualization of multi-omics data[J]. Nucleic Acids Research, 2018, 46(W1): W503-W509. DOI:10.1093/nar/gky466 |
| [18] |
ZAYKIN D V. Optimally weighted Z-test is a powerful method for combining probabilities in meta-analysis[J]. Journal of Evolutionary Biology, 2011, 24(8): 1836-1841. DOI:10.1111/j.1420-9101.2011.02297.x |
| [19] |
WHITLOCK M C. Combining probability from independent tests: the weighted Z-method is superior to Fisher's approach[J]. Journal of Evolutionary Biology, 2005, 18(5): 1368-1373. DOI:10.1111/j.1420-9101.2005.00917.x |
| [20] |
SATIZABAL C L, ADAMS H H H, HIBAR D P, et al. Genetic architecture of subcortical brain structures in 38, 851 individuals[J]. Nature Genetics, 2019, 51(11): 1624-1636. DOI:10.1038/s41588-019-0511-y |
| [21] |
WILLER C J, LI Y, ABECASIS G R. METAL: fast and efficient meta-analysis of genomewide association scans[J]. Bioinformatics, 2010, 26(17): 2190-2191. DOI:10.1093/bioinformatics/btq340 |
| [22] |
ADAMS H H H, HIBAR D P, CHOURAKI V, et al. Novel genetic loci underlying human intracranial volume identified through genome-wide association[J]. Nature Neuroscience, 2016, 19(12): 1569-1582. DOI:10.1038/nn.4398 |
| [23] |
CHANG C C, CHOW C C, TELLIER L C, et al. Second-generation PLINK: rising to the challenge of larger and richer datasets[J]. GigaScience, 2015, 4(1): 7. DOI:10.1186/s13742-015-0047-8 |
| [24] |
ZHANG K, CHANG S, GUO L, et al. I-GSEA4GWAS v2:a web server for functional analysis of SNPs in trait-associated pathways identified from genome-wide association study[J]. Protein & Cell, 2015, 6(3): 221-224. |
| [25] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017:quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45(D1): D362-D368. DOI:10.1093/nar/gkw937 |
| [26] |
SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Research, 2003, 13(11): 2498-2504. DOI:10.1101/gr.1239303 |
| [27] |
CHIN C H, CHEN S H, WU H H, et al. CytoHubba: identifying hub objects and sub-networks from complex interactome[J]. BMC Systems Biology, 2014, 8(Suppl 4): S11. DOI:10.1186/1752-0509-8-S4-S11 |
| [28] |
BADER G D, HOGUE C W. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4(1): 2. DOI:10.1186/1471-2105-4-2 |
| [29] |
HUMENIUK P, DUBIELA P, HOFFMANN-SOMMERGRUBER K. Dendritic cells and their role in allergy: uptake, proteolytic processing and presentation of allergens[J]. International Journal of Molecular Sciences, 2017, 18(7): 1491. DOI:10.3390/ijms18071491 |
| [30] |
ROCK K L, SHEN L. Cross-presentation: underlying mechanisms and role in immune surveillance[J]. Immunological Reviews, 2005, 207(1): 166-183. DOI:10.1111/j.0105-2896.2005.00301.x |
| [31] |
CHEN L, FLIES D B. Molecular mechanisms of T cell co-stimulation and co-inhibition[J]. Nature Reviews Immunology, 2013, 13(4): 227-242. DOI:10.1038/nri3405 |
| [32] |
MILNER J D. TCR signaling abnormalities in human Th2-associated atopic disease[J]. Frontiers in Immunology, 2018, 4(9): 719. |
| [33] |
ALEGRE M L, FRAUWIRTH K A, THOMPSON C B. T-cell regulation by CD28 and CTLA-4[J]. Nature Reviews Immunology, 2001, 1(3): 220-228. DOI:10.1038/35105024 |
| [34] |
许雪梅, 唐宗生, 李志鸿. T细胞CD28家族受体在哮喘发病中的作用[J]. 东南大学学报(医学版), 2016, 35(6): 1009-1013. XU X M, TANG Z S, LI Z H. The role of T cell CD28 family receptors in the pathogenesis of asthma[J]. Journal of Southeast University(Medical Science Edition), 2016, 35(6): 1009-1013. |
| [35] |
KELLY-WELCH A E. Interleukin-4 and Interleukin-13 signaling connections maps[J]. Science, 2003, 300(5625): 1527-1528. DOI:10.1126/science.1085458 |
| [36] |
秦庆华, 姚咏明. 转化生长因子β信号通路研究进展[J]. 医学研究杂志, 2012, 41(10): 5-9. QIN Q H, YAO Y M. Research progress of TGF-β signaling pathway[J]. Journal of Medical Research, 2012, 41(10): 5-9. |
| [37] |
TURNER J A, STEPHEN-VICTOR E, WANG S, et al. Regulatory T cell-derived TGF-β1 controls multiple checkpoints governing allergy and autoimmunity[J]. Immunity, 2020, 53(6): 1202-1214. DOI:10.1016/j.immuni.2020.10.002 |
| [38] |
胡东, 张荣波. 脂多糖对过敏性哮喘患者细胞因子分泌的影响[J]. 现代预防医学, 2007, 34(2): 237-239. HU D, ZHANG R B. Study on the effect of the lipopolysaccharide on secretory cytokine in allergic asthma patients in vitro[J]. Modern Preventive Medicine, 2007, 34(2): 237-239. |
| [39] |
姜勇, 龚小卫. MAPK信号转导通路对炎症反应的调控[J]. 生理学报, 2000, 52(4): 267-271. JINAG Y, GONG X W. Regulation of inflammatory responses by MAPK signal transduction pathways[J]. Acta Physiologica Sinica, 2000, 52(4): 267-271. |
| [40] |
CHOI J, KIM J R, KIM H, et al. The atopic dermatitis-like symptoms induced by MC903 were alleviated in JNK1 knockout mice[J]. Toxicological Sciences, 2013, 136(2): 443-449. DOI:10.1093/toxsci/kft215 |
| [41] |
DANDEKAR R D, KHAN M M. Regulation of ERK2 phosphorylation by histamine in splenocytes[J]. Immunopharmacology and Immunotoxicology, 2011, 33(2): 250-258. DOI:10.3109/08923973.2010.499913 |
| [42] |
HU P, NEBREDA A R, LIU Y, et al. p38α protein negatively regulates T helper type 2 responses by orchestrating multiple T cell receptor-associated signals[J]. Journal of Biological Chemistry, 2012, 287(40): 33215-33226. DOI:10.1074/jbc.M112.355594 |
| [43] |
PENDER S L, TICKLE S P, DOCHERTY A J, et al. A major role for matrix metalloproteinases in T cell injury in the gut[J]. The Journal of Immunology, 1997, 158(4): 1582-1590. |
| [44] |
DAHLEN B, SHUTE J, HOWARTH P. Immunohistochemical localisation of the matrix metalloproteinases MMP-3 and MMP-9 within the airways in asthma[J]. Thorax, 1999, 54(7): 590-596. DOI:10.1136/thx.54.7.590 |
| [45] |
MA L, BROWN M, KOGUT P, et al. Akt activation induces hypertrophy without contractile phenotypic maturation in airway smooth muscle[J]. American Journal of Physiology-Lung Cellular and Molecular Physiology, 2011, 300(5): L701-L709. DOI:10.1152/ajplung.00119.2009 |
| [46] |
崔庆新. 基于化学生物学的中药有效成分牛蒡子苷元等的作用靶点及体内分布研究[D]. 天津: 南开大学, 2014. CUI Q X. Target proteins and biodistribution research of arctigenin, an effective compound of Traditional Chinese Medicine based on chemical biology[D]. Tianjin: Nankai University, 2014. |
| [47] |
FANG R, CUI Q, SUN J, et al. PDK1/Akt/PDE4D axis identified as a target for asthma remedy synergistic with β2AR agonists by a natural agent arctigenin[J]. Allergy, 2015, 70(12): 1622-1632. DOI:10.1111/all.12763 |
| [48] |
STURM E M, PARZMAIR G P, RADNAI B, et al. Phosphoinositide-dependent protein kinase 1(PDK1) mediates potent inhibitory effects on eosinophils: Leukocyte signaling[J]. European Journal of Immunology, 2015, 45(5): 1548-1559. DOI:10.1002/eji.201445196 |
| [49] |
RANDHAWA V, BAGLER G. Identification of SRC as a potent drug target for asthma, using an integrative approach of protein interactome analysis and in silico drug discovery[J]. OMICS: A Journal of Integrative Biology, 2012, 16(10): 513-526. DOI:10.1089/omi.2011.0160 |
| [50] |
KUMAR SINGH P, KASHYAP A, SILAKARI O. Exploration of the therapeutic aspects of Lck: A kinase target in inflammatory mediated pathological conditions[J]. Biomedicine & Pharmacotherapy, 2018, 108(18): 1565-1571. |
| [51] |
ZUEHLKE A D, BEEBE K, NECKERS L, et al. Regulation and function of the human HSP90AA1 gene[J]. Gene, 2015, 570(1): 8-16. DOI:10.1016/j.gene.2015.06.018 |
| [52] |
陈晨. 基于网络药理学的玉屏风散有效成分改善小鼠接触性皮炎的机制研究[D]. 南京: 南京大学, 2017. CHEN C. The study on mechanism of Yupingfeng San's anti-inflammatory effect in contact dermatitis mice based on network pharmacology[D]. Nanjing: Nanjing University, 2017. |
| [53] |
姜盼盼, 王显红, 董昌宏, 等. JAK3在临床应用中的相关性研究及进展(综述)[J/OL]. 中国现代医学杂志, 2020: 1-7[2021-01-07]. http://kns.cnki.net/kcms/detail/43.1225.R.20200917.1703.011.html. JIANG P P, WANG X H, DONG C H, et al. Correlation and progress of JAK3 in clinical application[J/OL]. China Journal of Modern Medicine, 2020: 1-7[2021-01-07]. http://kns.cnki.net/kcms/detail/43.1225.R.20200917.1703.011.html. |
| [54] |
ALVES DE MEDEIROS A K, SPEECKAERT R, DESMET E, et al. JAK3 as an emerging target for topical treatment of inflammatory skin diseases[J]. PLoS One, 2016, 11(10): e0164080. DOI:10.1371/journal.pone.0164080 |
| [55] |
于孟斌, 杨予涛, 周晓巍, 等. PARP的生物学与病理学功能[J]. 中国生物化学与分子生物学报, 2008, 24(12): 1097-1102. YU M B, YANG Y T, ZHOU X W, et al. Biological and pathological functions of PARPs[J]. Chinese Journal of Biochemistry and Molecular Biology, 2008, 24(12): 1097-1102. |
| [56] |
ZAFFINI R, GOTTE G, MENEGAZZI M. Asthma and poly(ADP-ribose) polymerase inhibition: a new therapeutic approach[J]. Drug Design, Development and Therapy, 2018, 2(12): 281-293. |
2. Department of Psychology, University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Traditional Chinese Medicine, Beijing University of Chinese Medicine, Beijing 100029, China;
4. National Institute of Traditional Chinese Medicine Constitution and Preventive Medicine, Beijing University of Chinese Medicine, Beijing 100029, China
 2021, Vol. 38
2021, Vol. 38