文章信息
- 郭华, 徐流巍, 邢志美, 等.
- GUO Hua, XU Liuwei, XING Zhimei, et al.
- 基于板蓝根及其混伪品叶绿体基因组的mini-barcode开发及其定性能力的研究
- Development and identification of mini-barcode based on chloroplast genome of Isatis indigotica and its adulterants
- 天津中医药, 2023, 40(5): 662-667
- Tianjin Journal of Traditional Chinese Medicine, 2023, 40(5): 662-667
- http://dx.doi.org/10.11656/j.issn.1672-1519.2023.05.21
-
文章历史
- 收稿日期: 2023-02-01
2. 现代中医药海河实验室,天津 301617
板蓝根又名北板蓝根,《中国药典》[1]板蓝根的基原植物为十字花科植物菘蓝(Isatis indigotica),《中国植物志》将Isatis tinctoria作为菘蓝异名[2]。板蓝根是一味历史悠久、临床应用广泛的常用中草药,文化底蕴丰厚,早在《本草经集注》《本草图经》《三农纪》等均有记载[3]。《本草便读》记载板蓝根即靛青根,其功用性味与靛青叶同,入肝胃血分,能清热、解毒、辟疫、杀虫。
目前,医药市场中以板蓝根为主要原料的中西成药、中药饮片、兽药已超过2 000种,并且每年对板蓝根的需求量以15%的速度递增[4]。在实际应用中,板蓝根的使用一直较为混乱,常常将南板蓝根当作板蓝根使用[5],极易引起混淆[6-7],导致以假充真现象时有发生。《中国药典》南板蓝根的基原植物为爵床科植物马蓝Baphicacanthus cusia(Nees)Bremek,《中国植物志》将Strobilanthes cusia作为异名[8]。传统的板蓝根鉴定方法[9]依赖长期经验,而且环境或人为因素的干扰会影响植物特征,这都给准确鉴定带来一定难度。分子鉴定技术不受上述问题的影响,其中,DNA mini-barcode利用物种的、足够变异的、相对较短的DNA序列进行快速、准确获得物种信息[10],是传统中药鉴定方法(性状鉴定、显微鉴定和理化鉴定)的重要补充。
相较于传统的DNA条形码,叶绿体全基因组具有分子量小,多拷贝和进化速率适中等特征,现已广泛应用于植物的系统发育和物种鉴定中[11-13]。综上,研究利用GenBank数据库中已经公布的板蓝根和南板蓝根的叶绿体基因组序列开发mini-barcode,采用试剂盒法提取板蓝根药材总DNA分别利用通用ITS2引物和本实验设计引物进行聚合酶链式反应(PCR)和测序,验证开发的mini-barcode是否可用于板蓝根的定性研究,为板蓝根药材和含板蓝根的中药制剂的质量控制提供参考。
1 材料与方法 1.1 实验材料从美国国立生物技术信息中心(NCBI)的GenBank数据库下载板蓝根,南板蓝根的ITS2序列和叶绿体基因组序列。分别收集来自甘肃太和和民乐的板蓝根样品,共分为6个批次,18份样品。样本来源信息见表 1。
实验仪器:PCR仪(伯乐生命医学产品有限公司,型号:T100),超净工作台(上海智城分析仪器制造有限公司,型号:ZHJH-C1112B),Nano Drop分光光度计(赛默飞世尔科技公司,型号:TM),电泳仪(北京君意东方电泳设备有限公司,型号:JY300C),千分之一电子天平(常州市天之平仪器设备有限公司,型号:EL-220A),水平电泳槽(上海君易科学仪器有限公司,型号:HE-90),荧光定量仪(赛默飞世尔科技公司,型号:Qubit 2.0),紫外分析仪(上海嘉鹏科技有限公司,型号:ZF-6)。
实验试剂:植物基因组DNA提取试剂盒,无水乙醇,巯基乙醇,氯仿,Tks Gflex DNA聚合酶,Du Red核酸染料,DL 500 DNA marker,DL 2000 DNA marker,6×loading buffer。
1.3 实验方法 1.3.1 DNA提取板蓝根清水冲洗,使用75%乙醇擦拭表面,晾干。分别用液氮研磨板蓝根药材30 mg装入1.5 mL离心管。利用天根植物基因组DNA提取试剂盒(DP305)按照说明书进行DNA提取。提取过程中,离心管水浴锅65 ℃加热1 h,设立空管阴性对照。采用Nanodrop 2000微量分光光度计测定DNA质量浓度。
1.3.2 DNA mini-barcode开发已下载的板蓝根和南板蓝根的叶绿体基因组序列,导入PhyloSuite v1.2.1[14]软件提取其共有蛋白编码基因并分别对齐,后续使用DnaSP 6[15]软件进行核苷酸多态性分析以筛选出候选DNA mini-barcode。随后使用Geneious[16]软件对筛选出的候选片段多序列比对,使用“Primer Design”功能选择合适的长度进行引物设计即得开发的DNA分子标记,参数为默认值。使用Oligo 7[17]对引物的物理化学性质进行评估。
1.3.3 遗传距离计算将候选mini-barcode对应的所有序列导入MEGA[18],利用K2P双参数模型,计算遗传距离以判断板蓝根与南板蓝根的该扩增子序列是否在遗传差异。
1.3.4 PCR扩增及测序分别使用通用ITS2引物(正向:ATGCGATACTTGGTGTGAAT,反向:GACGCTTCTCCAGACTACAAT)和本实验设计accD引物对提取的每份相同的样本DNA进行PCR扩增,分别设立负管阴性对照。PCR反应体系包括Tks Gflex DNA Polymerase 1 μL、2×Gflex Buffer 25 μL、引物(10 μmol/L)各1 μL、模板DNA 2 μL,加水补足至50 μL。ITS2序列的PCR反应条件:98 ℃ 1min,98 ℃ 5 s,50 ℃ 15 s,94 ℃ 20 s,循环35次,72 ℃ 5 min。accD序列的PCR反应条件:98 ℃ 1 min,98 ℃ 5 s,56 ℃ 15 s,94 ℃ 20 s,循环35次,72 ℃ 5min。PCR反应结束后,对扩增产物进行2%琼脂糖凝胶电泳。最终由北京华大测序公司完成双端测序。
1.3.5 数据处理测序所得的峰图文件和序列文件利用Geneious软件采用de novo拼接方法得到一致性序列,同时去除低质量序列及引物序列。使用MAFFT[19]分别得到的最终序列进行比对和矫正。分别将本研究获得ITS2序列和accD扩增叶绿体基因组区域结合已发表的相应序列构建系统发育树,观察板蓝根序列的分布情况,设置bootstrap值为1 000。
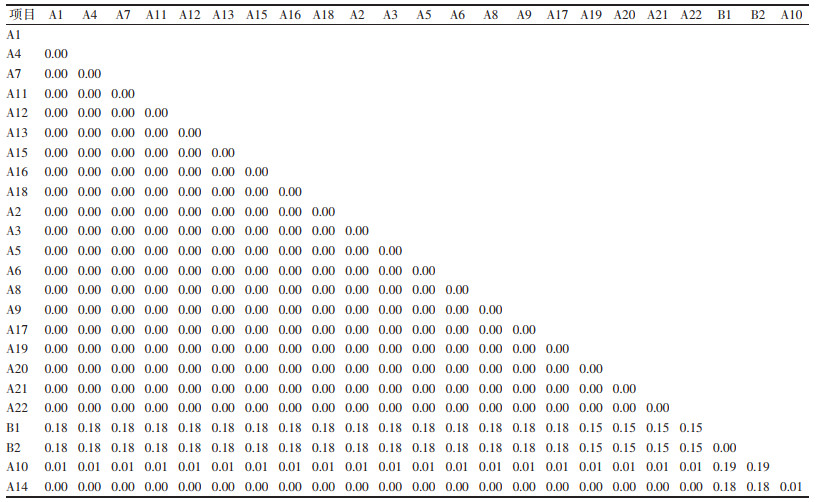
2 结果 2.1 mini-barcode开发计算板蓝根、南板蓝根的叶绿体基因组序列的蛋白编码基因区域的核酸多态性(Pi值),筛选出最优的DNA分子标记候选区域,见图 1。ccsA的Pi值最高(0.290 67),其次是accD(0.275 36)、ndhF(0.226 07)、ycf1(0.217 14)、rpoC2(0.21)、rpl22(0.205 71)。针对这些高变区进行mini-barcode的引物设计。考虑条形码长度和引物设计的原则,最终选择accD作为DNA候选分子标记区域用来设计引物。使用Oligo 7软件对在accD区域成功设计的引物的物理化学性质评估并确定最终引物序列,具体引物信息见表 2。
|
| 图 1 核酸变异度分析 Fig. 1 Nucleic acid variability analysis |
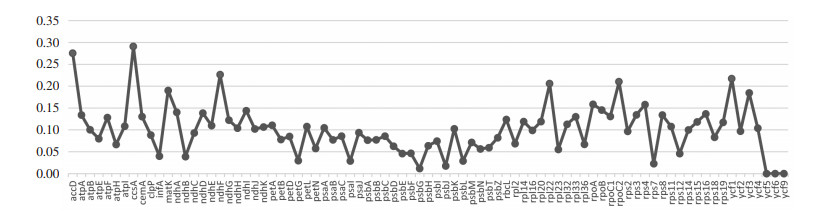
核酸检测仪测定结果显示所有样品DNA平均质量浓度为14.7 ng/μL,D260 nm/D280 nm为1.97。所有扩增产物均有条带且符合预期扩增长度,且空管(K)和负管(F)无污染,见图 2。
|
| 注:左为ITS2序列,右为mini-barcode序列,K为空管对照,F为负管对照。 图 2 凝胶电泳图 Fig. 2 The gel electrophoresis graph |
种内种间遗传距离结果见表 3。板蓝根的种内最大遗传距离为0.01,与南板蓝根的种间最小遗传距离为0.15,种内最大遗传距离小于种间最小遗传距离,故mini-barbode序列可将板蓝根与南板蓝根之间可以分开。见表 3。
板蓝根药材的ITS2扩增子序列长度均为416~420 bp,而mini-barcode的扩增子序列长度均为165 bp。本研究所得ITS2和accD序列结合已发表的相关序列构建系统发育树,结果见图 3。mini-barcode序列的系统发育树的聚类分析结果表明板蓝根与南板蓝根聚在不同枝上,能够将板蓝根及南板蓝根有效区分。系统发育树同时也表明DNA mini-barcode与ITS2的物种分辨率相当,说明本研究的DNA mini-barcode适用于DNA降解严重样品或混合样品的鉴定研究。
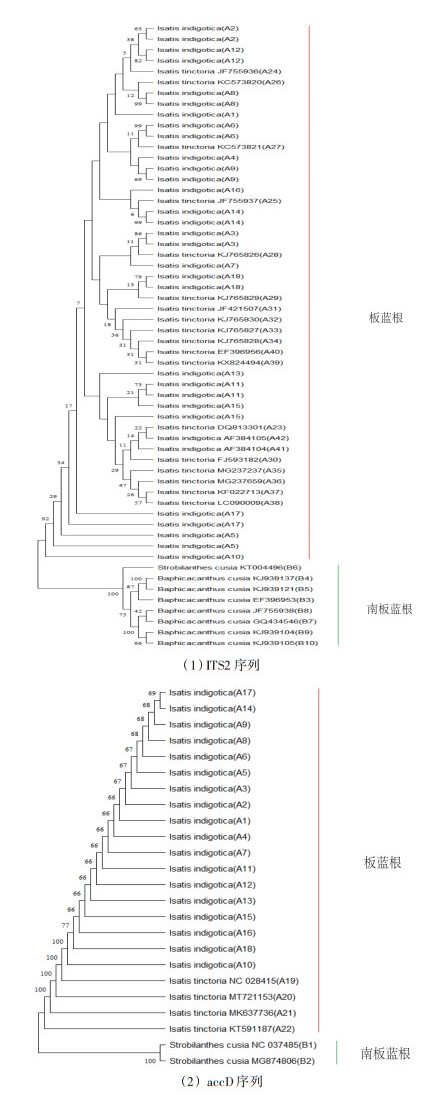
|
| 图 3 系统发育树 Fig. 3 Phylogenetic tree |
研究基于板蓝根及其混伪品叶绿体基因组的开发mini-barcode,采用试剂盒法提取板蓝根药材总DNA分别利用通用ITS2引物和本实验设计引物进行PCR和测序。通过遗传距离和测序数据分析,该分子标记能被成功扩增并实现鉴定,为实现板蓝根药材及含板蓝根的中成药等可能存在DNA降解的样本或混合样本的真伪鉴别奠定基础。
4 讨论在人类几千年的文明发展史中,植物一直扮演着重要的角色。凭借着丰富的植物物种资源中国孕育了宝贵的中医药文化。中药资源是中医药文化赖以生存和发展的重要物质基础[20]。传统的鉴定方法存在局限,DNA条形码技术在该领域广泛和深入的研究和应用,是传统鉴定方法的重要补充,也是未来发展的必然趋势。
黄志海等[21]利用ITS2条形码对单株植物板蓝根与南板蓝根成功鉴定。应用ITS2通用引物对中成药中板蓝根进行扩增鉴定时,ITS2通用引物对于中成药中板蓝根扩增效率会受到影响,进而影响鉴定。由此可见,笔者开发的mini-barcode对药材及含板蓝根的中成药的鉴定具有一定的优势。DNA mini-barcode长度较短(通常小于200 bp),更容易扩增成功,与通用的ITS2序列相比,实际应用范围更广[22-25]。事实上,药材从采收到加工的任何一个过程都极有可能影响样本DNA质量,导致鉴定失败。研究发现mini-barcode是鉴定中药材及中成药的更佳选择。通常,在选择mini-barcode时需要满足3个条件:1)包含显著的种间遗传变异。2)存在稳定的种内遗传变异。3)具有保守的侧翼序列。蛋白编码基因是常用的分子进化研究标记。其具有保守区和可变区,且一般为单拷贝基因,不存在由种内序列异质性引起的偏差。在核苷酸序列水平上,由于蛋白编码基因允许密码子的第1位和第3位发生同义突变,不同物种的蛋白编码基因序列差异显著,有利于物种鉴定。本实验基于板蓝根及其混伪品的叶绿体基因组序列筛选出高变区accD,其包含了可区分板蓝根及其混伪品的变异位点,且变异位点两端序列保守,因此在accD区域成功开发出DNA分子标记。实验首次实现了对含有板蓝根的中成药的定性鉴别,为其他分类复杂、化学成分相似的中药材和中成药鉴定提供了研究实例。
| [1] |
国家药典委员会. 中华人民共和国药典-一部: 2020年版[M]. 北京: 中国医药科技出版社, 2020: 215-216. Chinese Pharmacopoeia Commission. Pharmacopoeia of People's Republic of China.Part 1:2020 edition[M]. Beijing: China Medical Science Press, 2020: 215-216. |
| [2] |
中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 1987: 64. Editorial Committee of the Flora of China, Chinese Academy of Sciences. Flora reipublicae popularis sinicae[M]. Beijing: Science Press, 1987: 64. |
| [3] |
王艺涵, 金艳, 陈周全, 等. 蓝草类药材的本草考证[J]. 中国中药杂志, 2020, 45(23): 5819-5828. WANG Y H, JIN Y, CHEN Z Q, et al. Textual research on medicinal blue herbs[J]. China Journal of Chinese Materia Medica, 2020, 45(23): 5819-5828. |
| [4] |
张明生, 宋立东, 高文丽, 等. 吉林省板蓝根垄作栽培技术[J]. 中国园艺文摘, 2010, 26(12): 184-185. ZHANG M S, SONG L D, GAO W L, et al. Ridge cultivation techniques of Radix isatidis in Jilin Province[J]. Chinese Horticulture Abstracts, 2010, 26(12): 184-185. |
| [5] |
刘保财, 陈菁瑛, 张武君, 等. 马蓝生产技术规范化流程与思考[J]. 福建农业科技, 2021, 52(9): 64-68. LIU B C, CHEN J Y, ZHANG W J, et al. Standardization process of production technology for Strobilanthes cusia[J]. Fujian Agricultural Science and Technology, 2021, 52(9): 64-68. |
| [6] |
唐璇. 板蓝根药用史考[J]. 环球中医药, 2014, 7(11): 869-871. TANG X. Textual research on medicinal history of Radix Isatidis[J]. Global Traditional Chinese Medicine, 2014, 7(11): 869-871. |
| [7] |
申琼琪, 侯惠婵, 栗建明, 等. 板蓝根与南板蓝根及其伪品的比较鉴别[J]. 中国医药工业杂志, 2014, 45(1): 31-34. SHEN Q Q, HOU H C, LI J M, et al. Comparison and identification of Radix isatidis, baphicacanthis cusiae rhizoma and its adulterants[J]. Chinese Journal of Pharmaceuticals, 2014, 45(1): 31-34. |
| [8] |
中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 2002: 113. Editorial Committee of the Flora of China, Chinese Academy of Sciences. Flora reipublicae popularis sinicae[M]. Beijing: Science Press, 2002: 113. |
| [9] |
罗晓铮, 代丽萍, 刘孟奇, 等. 菘蓝叶和根的解剖及生物碱的组织化学定位研究[J]. 河南农业科学, 2016, 45(10): 119-122. LUO X Z, DAI L P, LIU M Q, et al. Anatomy and histochemical localization of alkaloids in the leaf and root of Isatis indigotica fort[J]. Journal of Henan Agricultural Sciences, 2016, 45(10): 119-122. |
| [10] |
HAJIBABAEI M, SMITH M A, JANZEN D H, et al. A minimalist barcode can identify a specimen whose DNA is degraded[J]. Molecular Ecology Notes, 2006, 6(4): 959-964. |
| [11] |
赵玉芬. 叶绿体基因组在植物学研究中的应用进展[J]. 生物学教学, 2022, 47(3): 83-85. ZHAO Y F. Application progress of chloroplast genome in botanical research[J]. Biology Teaching, 2022, 47(3): 83-85. |
| [12] |
LI L D, JIANG Y, LIU Y Y, et al. The large single-copy (LSC) region functions as a highly effective and efficient molecular marker for accurate authentication of medicinal Dendrobium species[J]. Acta Pharmaceutica Sinica B, 2020, 10(10): 1989-2001. |
| [13] |
ZHANG Y, ZHANG A H, LI X M, et al. The role of chloroplast gene expression in plant responses to environmental stress[J]. International Journal of Molecular Sciences, 2020, 21(17): 6082. |
| [14] |
ZHANG D, GAO F L, JAKOVLIĆ I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources, 2020, 20(1): 348-355. |
| [15] |
ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6:DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution, 2017, 34(12): 3299-3302. |
| [16] |
KEARSE M, MOIR R, WILSON A, et al. Geneious Basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28(12): 1647-1649. |
| [17] |
HAUGLAND R A, HECKMAN J L. Identification of putative sequence specific PCR primers for detection of the toxigenic fungal speciesStachybotrys chartarum[J]. Molecular and Cellular Probes, 1998, 12(6): 387-396. |
| [18] |
KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. |
| [19] |
KATOH K, MISAWA K, KUMA K, et al. MAFFT: A novel method for rapid multiple sequence alignment based on fast Fourier transform[J]. Nucleic Acids Research, 2002, 30(14): 3059-3066. |
| [20] |
刘文龙, 赵靖, 李原华, 等. 中药宏观质量的评价与控制理论体系的建立与应用研究[J]. 中草药, 2021, 52(3): 613-620. LIU W L, ZHAO J, LI Y H, et al. Study on establishment and application of evaluation and control theory system of holistic quality of traditional Chinese medicine[J]. Chinese Traditional and Herbal Drugs, 2021, 52(3): 613-620. |
| [21] |
黄志海, 丘小惠, 宫璐, 等. 板蓝根与南板蓝根及其混淆品的ITS2条形码鉴定[J]. 中药材, 2017, 40(1): 50-53. HUANG Z H, QIU X H, GONG L, et al. Identification of Isatis tinctoria, Baphicacanthus cusia and thier adulterants with ITS2 barcode[J]. Journal of Chinese Medicinal Materials, 2017, 40(1): 50-53. |
| [22] |
GALAN M, PAGÈS M, COSSON J F. Next-generation sequencing for rodent barcoding: species identification from fresh, degraded and environmental samples[J]. PLoS One, 2012, 7(11): e48374. |
| [23] |
PARVEEN I, TECHEN N, KHAN I A. Identification of species in the aromatic spice family apiaceae using DNA mini-barcodes[J]. Planta Medica, 2019, 85(2): 139-144. |
| [24] |
PANDIT R, TRAVADI T, SHARMA S, et al. DNA meta-barcoding using rbcL based mini-barcode revealed presence of unspecified plant species in ayurvedic polyherbal formulations[J]. Phytochemical Analysis, 2021, 32(5): 804-810. |
| [25] |
FRANÇOSO E, ARIAS M C. Cytochrome c oxidase I primers for corbiculate bees: DNA barcode and mini-barcode[J]. Molecular Ecology Resources, 2013, 13(5): 844-850. |
2. Haihe Laboratory of Modern Chinese Medicine, Tianjin 301617, China
 2023, Vol. 40
2023, Vol. 40