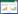文章信息
- 刘金杰, 黄湘龙, 宋柯妍, 等.
- LIU Jinjie, HUANG Xianglong, SONG Keyan, et al.
- 基于生物信息学结合网络药理学探讨党参改善低氧适应的作用机制
- Exploring the mechanism by which Codonopsis Radix improves hypoxia adaptation based on bioinformatics and network pharmacology
- 天津中医药, 2025, 42(2): 204-215
- Tianjin Journal of Traditional Chinese Medicine, 2025, 42(2): 204-215
- http://dx.doi.org/10.11656/j.issn.1672-1519.2025.02.12
-
文章历史
- 收稿日期: 2024-08-27
2. 组分中药国家重点实验室, 天津 301617;
3. 现代中药创制全国重点实验室, 天津 301617;
4. 方剂学教育部重点实验室, 天津 301617
低氧适应是指当组织细胞氧含量低于正常水平时,组织细胞的代谢、功能和形态结构发生异常变化。为了适应缺氧,机体发生一系列局部性和系统性的改变以降低缺氧的影响[1-2]。人体的低氧适应机制极其复杂,在氧气缺乏的情况下,人体细胞一系列信号通路被激活,主要涉及缺氧诱导因子、能量代谢途径以及细胞应激通路等,从而诱导细胞对缺氧的应激反应。到目前为止,研究最充分的适应机制是缺氧诱导因子-1(HIF-1)的稳定化,这一研究成果为细胞低氧适应机制奠定了基础,并获得了2019年诺贝尔生理学或医学奖[3]。HIF通路的发现,明确了细胞如何感知氧的变化、介导下游的信号通路,为防治低氧相关疾病提供了新靶点。HIF-1α是当前防治缺氧相关疾病的首选药物靶标,临床试验中的大多数药物都是围绕调控HIF-1α mRNA的转录水平、调控HIF-1α蛋白的稳定性、改变HIF-1α蛋白的酶活性等开展设计[4]。
党参为桔梗科植物党参Codonopsis pilosula(Franch)Nannf、素花党参Codonopsis pilosula Nannf.var.modesta(Nannf.)L.T.Shen或川党参Codonopsis tangshen Oliv的干燥根,具有健脾益肺,养血生津的功效[5],是中国常用的一种传统补益类中药。其主要含有糖类、生物碱类、聚炔类、苷类及萜类等成分,具有抗缺氧、抗疲劳、抗应激、提高人体免疫力、调节血糖、改善造血机能、延缓衰老、保护胃黏膜及抗溃疡等多种药理作用[6-7],但提高人体低氧适应的药理作用尚未明确。
因此,文章基于网络药理学结合GEO数据库等生物信息学技术,通过两种机器学习的方法深度挖掘有关低氧适应的核心基因,总结探讨党参改善低氧适应机制的核心通路。结合数据库预测及分子对接探讨党参改善低氧适应的分子机制,以期为临床防治相关疾病提供思路与参考。
1 材料与方法 1.1 党参化学成分数据库的建立及活性成分的筛选通过综合数据收集党参的主要活性成分[8]。首先,以“Codonopsis Radix”为关键词检索TCMSP、BATMAN-TCM和PubChem专业药物数据库[9-10],并以口服生物利用度(OB)≥30%、类药性(DL)≥0.18为筛选标准,筛选党参主要活性成分;并通过CNKI和Pubmed报道的活性成分进行补充。
1.2 党参活性成分靶点的预测相似性集成方法(SEA)基于蛋白质配体之间的化学相似性来关联蛋白质,可以用于快速搜索大型化合物数据库,并寻找药物潜在靶点[11]。SuperPred分析平台可以将类药物化合物的化学相似性与分子靶标基于相似性原理的方法联系起来,预测总准确度可达到75.1%[12]。该研究通过SEA(https://sea.docking.org)和SuperPred分析平台(https://prediction.charite.de)查找党参主要活性成分对应的潜在作用靶点,并通过UniProt数据库进行标准化处理,最终得到党参活性成分靶点。
1.3 基因芯片数据获取和差异表达基因的筛选从基因表达数据库(GEO)中获取GSE244830数据集。GSE244830数据集中包含有6例常氧环境下C57BL/6J小鼠基因表达谱以及6例缺氧环境(10% O2)下小鼠基因表达谱,使用R软件对每组芯片数据进行标准化和log2的转换。使用“limmar”包鉴定差异表达基因(DEGs),以|log2FC|≥1.0且P<0.05为筛选条件。使用“pheatmap”和“ggVolcano”软件包绘制可视化热图和火山图,以显示重要DEGs表达。对DEGs进行基因集富集分析(GSEA),探究差异基因富集的显著表型。
1.4 低氧适应潜在靶点数据集建立在GeneCards数据库中,以“hypoxia adaption”为检索词,检索与低氧适应相关的基因,随后导入UniProt数据库进行标准化处理,并与上述DEGs汇总,建立低氧适应潜在靶点数据集。
1.5 药物-疾病共同作用靶点的获取及网络的构建将党参活性成分靶点与低氧适应潜在靶点数据集取交集,获得药物-疾病共同作用靶点。运用Cytoscape 3.7.1软件绘制并构建“党参活性成分-低氧适应共同作用靶点”的关系网络。
1.6 蛋白互相作用(PPI)网络构建与潜在核心靶点的筛选将“党参活性成分-低氧适应共同作用靶点”网络,导入String数据库,选择medium confidence=0.4作为最小互作分数进行PPI网络分析;运用Cytoscape 3.7.1以连接度(Degree)、紧密度(CC)以及介度(BC)的中位数为筛选标准,对其进行拓扑学参数分析,重新构建核心靶点的PPI网络图,即通过网络药理学筛选得到党参-低氧适应潜在核心靶点。
1.7 机器学习对潜在核心靶点的筛选最小绝对收缩和选择算子(LASSO)通过在常用的多元线性回归中添加惩罚函数(λ),不断压缩系数,从而达到精简模型的目的,其优势在于可以利用较少样本量,高效筛选较多变量[13]。支持向量机(SVE)是一种在机器学习领域中用于分类和回归分析的强大算法,在R软件中调用“e1071、kernlab和caret”R包来进行SVE计算,可通过训练样本,不断迭代,最后选出需要的特征数,识别可能的关键基因[14]。该研究采用LASSO回归模型和SVE机器学习方法,进一步寻找党参活性成分与低氧适应的潜在作用靶点。将机器学习得到的潜在核心靶点与上述网络药理学筛选的核心靶点汇总,确定为党参提高低氧适应的核心靶点。
1.8 核心靶点的验证利用R软件对核心基因进行训练集GSE244830的表达水平验证。并引用GSE186474作为验证集,进一步验证低氧下核心靶点的表达水平;iHypoxia数据库[15]收录了大量针对缺氧反应机制的蛋白靶点,利用iHypoxia数据库对核心靶点再次进行验证,以提高靶点预测的准确性。
1.9 GO功能富集分析和KEGG通路富集分析采用clusterProfiler R软件包对核心基因进行GO分析,探索其潜在的生物学功能;KEGG通路富集分析,探索其生物学途径和潜在信号通路。
1.10 分子对接从RCSB PDB数据库和PubChem数据库分别获得低氧适应潜在核心靶点蛋白结构以及党参活性成分结构。借助Discovery Studio 2020客户端进行分子对接,对蛋白进行去除杂原子及多余水分子,加氢,计算电场强度,力场最小化等处理,并识别蛋白结合位点,锁定对接口袋的位置和大小;小分子处理则通过“Prepare Ligands”选项来执行产生三维结构及异构体,优化力场等操作。使用CDOCKER对接方式进行对接及结合能预测,结合能<0 kcal/mol表明配体小分子化合物可以与蛋白受体进行自发结合;结合能≤-5 kcal/mol表明配体小分子化合物与蛋白受体具有良好的结合活性。
2 结果 2.1 党参的主要活性成分及作用靶点预测以OB≥30%、DL≥0.18为筛选标准,对公共数据库查找的靶点进行筛选,共得到党参活性成分15种;结合文献报道,增加党参苷I(Tangshenoside I)、党参炔苷(Lobetyolin)、党参碱(Codonopsine)和紫丁香苷(Syringin)4个药理活性成分[16-17],共得到活性成分19个,见表 1;通过SEA和SuperPred分析平台查找,共得到活性成分靶点338个。
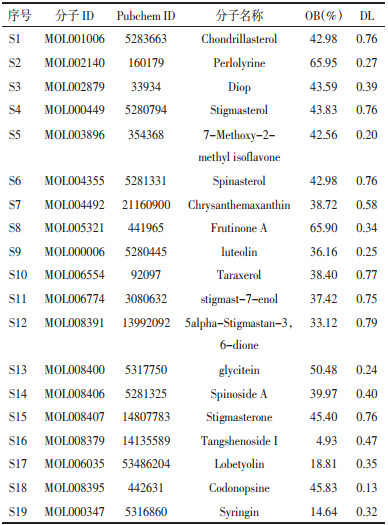
GSE244830数据集中共有1 306个基因被鉴定为低氧适应相关DEGs,其中上调基因475个,下调基因831个,火山图如图 1A所示,红色和绿色节点分别表示上调和下调基因,差异不显著基因为灰色。热图展示了差异显著的排名前200的基因,如图 1B所示。GSEA富集结果显示,DEGs主要与有丝分裂细胞周期相变、细胞器组装等生物学过程相关,见图 1C。
|
| 注:图A,GSE244830芯片DEGs火山图;图B,GSE244830芯片DEGs热图;图C,GSE244830芯片DEGs的GSEA富集结果。 图 1 GSE244830芯片中DEGs筛选 Fig. 1 Screening of DEGs based on GSE244830 microarray |
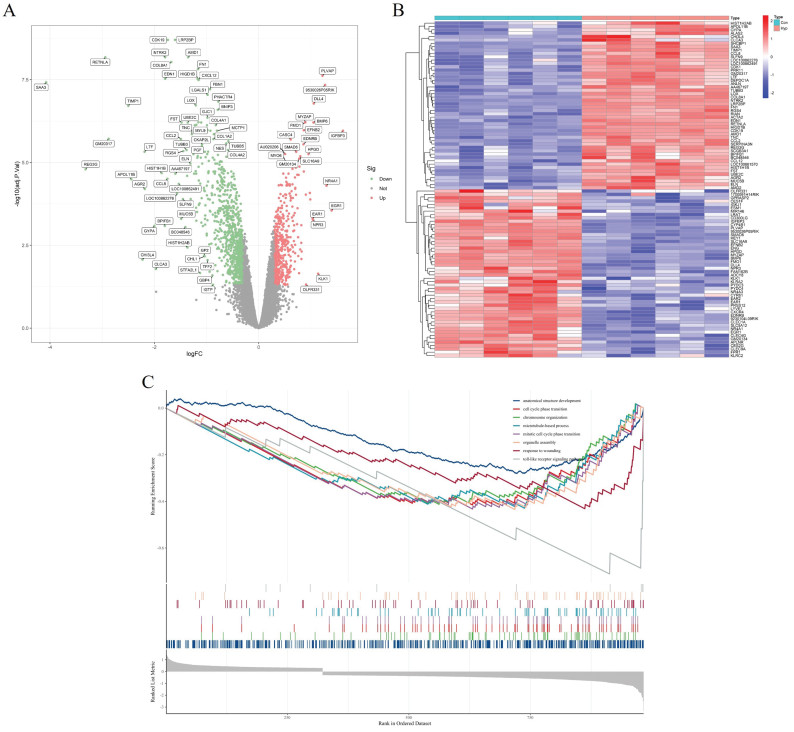
通过GeneCard数据库获得低氧适应相关靶点5 815个。将其与上述基因芯片筛选得到的差异基因进行交集,得到核心缺氧相关靶点476个,基于此建立低氧适应潜在靶点数据集,见图 2。
|
| 图 2 低氧适应潜在靶点数据集 Fig. 2 Potential target dataset for hypoxia adaptation |
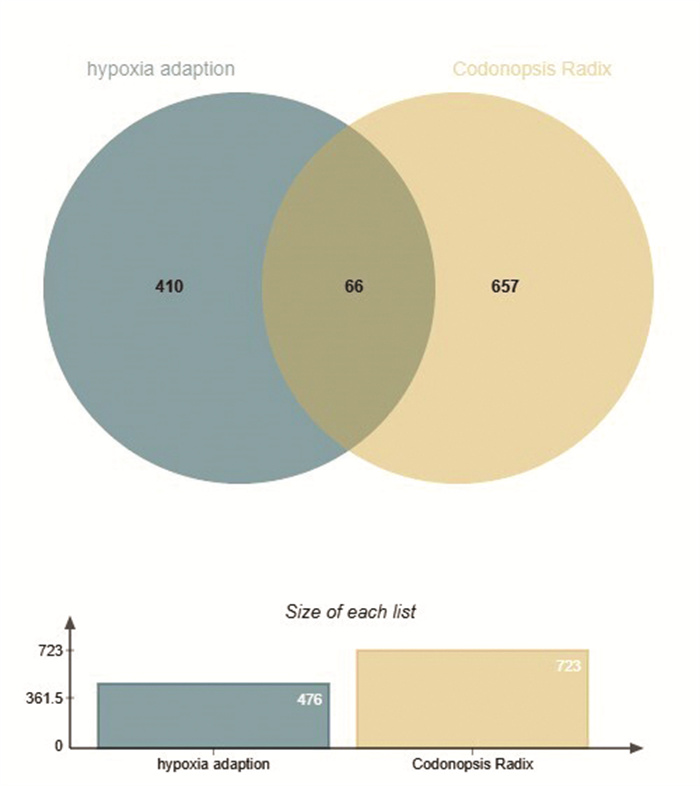
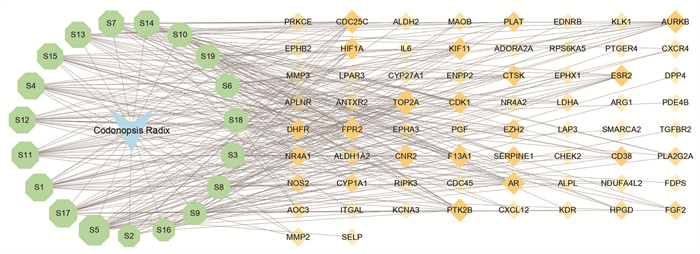
党参(Codonopsis Radix)与低氧适应(hypoxia adaption)共同靶点有66个,见图 3。将其进行可视化分析,见图 4。在网络中共85个节点(其中包括19个活性成分节点和66个靶点节点,六边形节点代表药物成分,菱形节点代表共同作用靶点)和291条边。
|
| 图 3 药物-疾病共同作用靶点 Fig. 3 Common interaction targets from drug and disease |
|
| 图 4 党参活性成分-低氧适应共同作用靶点网络 Fig. 4 Common interaction targets network of Codonopsis Radix active components and hypoxia adaptation |
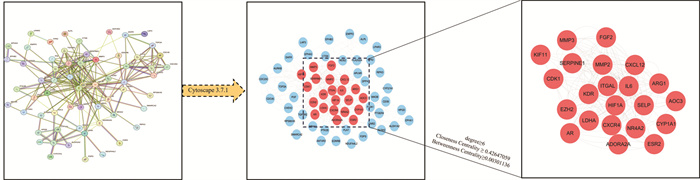
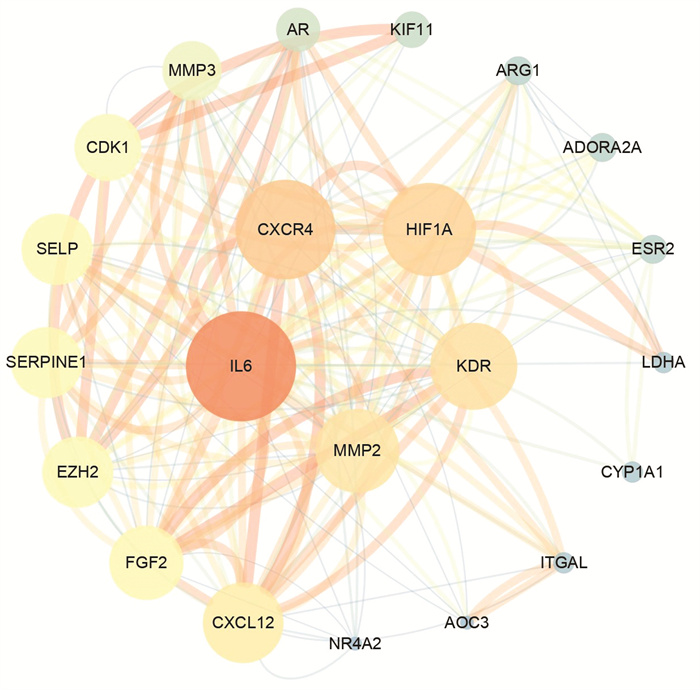
将66个共同作用靶点导入String数据库构建PPI网络,随后将其导入Cytoscape 3.7.1,对PPI网络进行可视化,并利用Network Analyzer程序计算核心靶点网络拓扑学参数,以Degree≥6、CC≥0.42647059以及BC≥0.00301136拓扑学参数条件进行筛选(中位数筛选),筛选得到22个核心靶点,如图 5所示;并根据靶点的degree值、combined score值正相关最终筛选得到排名前5的靶点分别是白细胞介素6(IL-6)、趋化因子受体(CXCR4)、缺氧诱导因子1α(HIF1A)、激酶插入域受体(KDR)和基质金属蛋白酶2(MMP2),如图 6所示。
|
| 图 5 党参-低氧适应靶点蛋白PPI筛选 Fig. 5 PPI screening of hypoxia adaptation target protein from Codonopsis Radix and hypoxia adaptation |
|
| 图 6 党参-低氧适应核心靶点蛋白PPI网格图 Fig. 6 PPI network grid of hypoxia adaptation core target protein from Codonopsis Radix and hypoxia adaptation |
LASSO回归分析得到9个潜在核心靶点基因,SVE模型筛选得到22个核心潜在靶点,如图 7A-C。通过两种机器学习最终确定3个党参抗缺氧的共同靶点:胎盘生长因子(PGF)、细胞周期蛋白依赖激酶1(CDK1)和KDR,见图 7D。
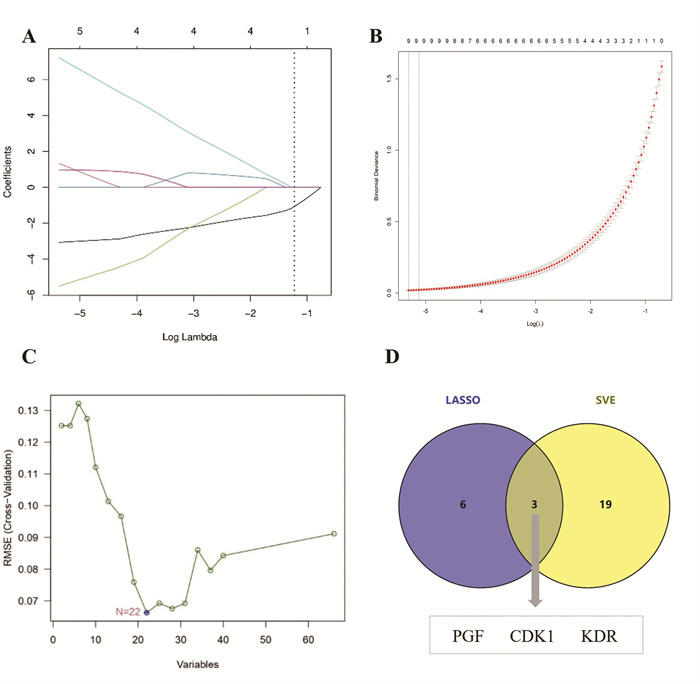
|
| 注:图A,LASSO回归模型中6个变量的系数曲线;图B,LASSO回归模型中转向参数(λ)选择的十重交叉验证;图C,基于22个特征基因的SVM最佳均方根误差(RMSE);图D,通过LASSO回归模型筛选出9个核心靶点和基于SVM筛选得到的22个核心靶点,取交集后得到3个共有核心靶点。 图 7 基于机器学习方法的核心靶点筛选 Fig. 7 Core target selection based on machine learning methods |
将网络药理学筛选排名前5的核心靶点(IL6、CXCR4、HIF1A、KDR和MMP2)和机器学习筛选得到靶点(PGF、CDK1和KDR)确定为核心靶点,并对其分别在训练集GSE244830和验证集GSE186474进行表达水平验证,发现7个核心基因在缺氧环境下均差异性表达,见图 8。随后采用iHypoxia数据库对其进行再次验证,发现7个核心靶点均被收录,由此说明筛选得到的7个核心靶点与低氧适应机制密切相关,同时进一步说明其可能为党参改善低氧适应的核心作用靶点。
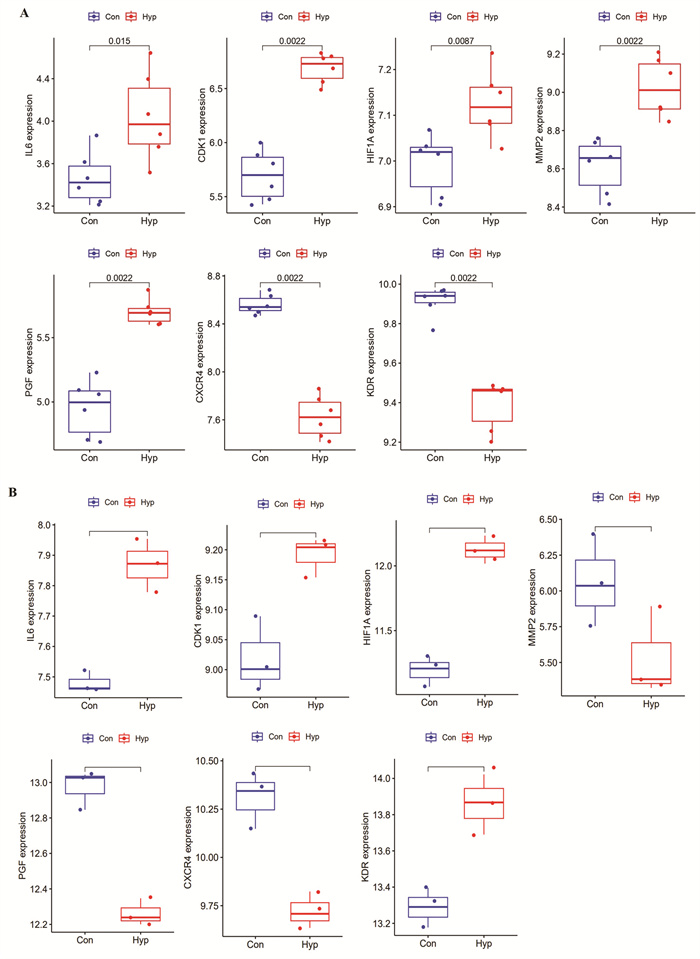
|
| 注:图A,GSE244830数据集核心靶点表达水平;图B,GSE186474数据集核心靶点表达水平。 图 8 核心基因的验证 Fig. 8 Verification of core genes |
GO富集分析得到BP相关条目31条、CC相关条目3条及MF相关条目6条(P<0.05),见图 9。结果显示,党参提高低氧适应的BP主要富集在对缺氧的反应、血管生成、内皮细胞增殖及分化等方面,CC主要富集在锚定连接点、细胞外区域和早期内体等方面。MF主要富集在Hsp90蛋白结合、共受体活性和生长因子活性等方面。
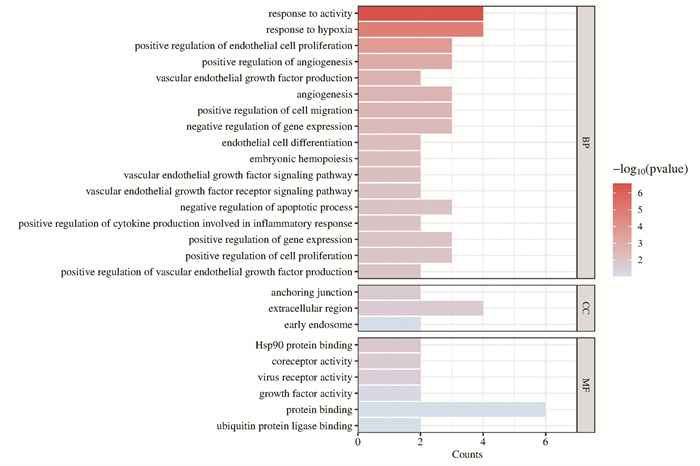
|
| 图 9 核心靶点的GO富集分析 Fig. 9 GO enrichment analysis of core targets |
KEGG信号通路分析得到20条相关信号通路(P<0.05)。其中与低氧适应相关的通路主要包括磷脂酰肌醇-3-激酶(PI3K)-蛋白激酶B(Akt)信号通路和HIF-1信号通路,见图 10。
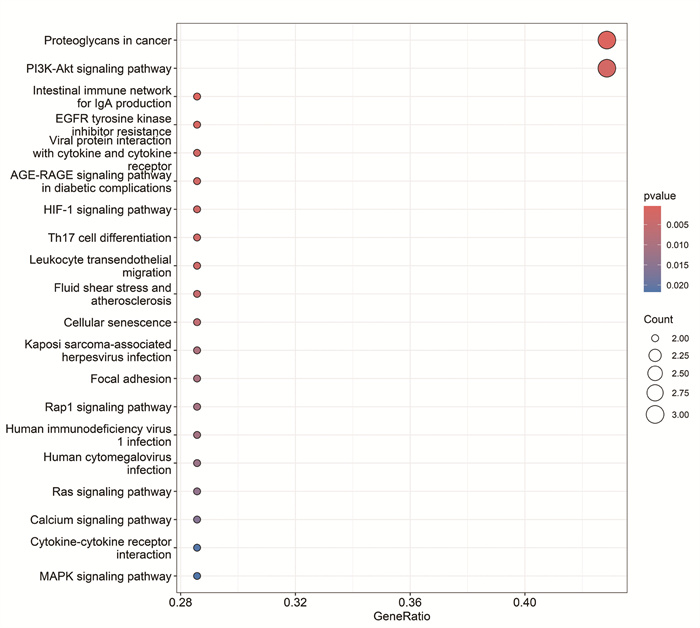
|
| 图 10 核心靶点的KEGG通路富集分析 Fig. 10 KEGG pathway enrichment analysis of core targets |
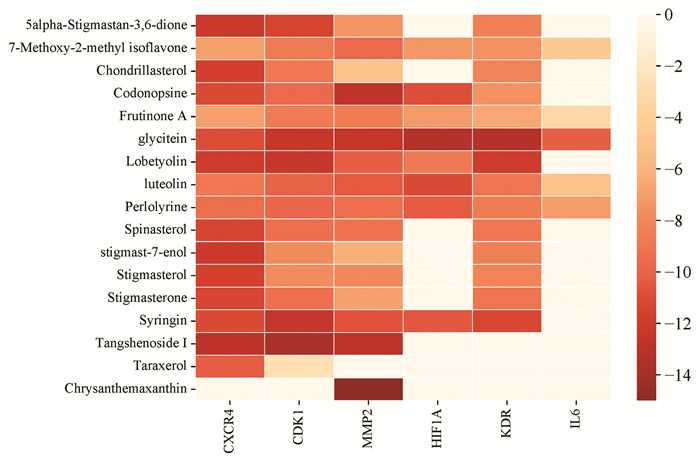
利用Discovery Studio对核心靶点HIF1A(PDB ID:1H2K)、KDR(PDB ID:1Y6A)、MMP2(PDB ID:7XJO)、PGF(PDB ID:1FZV)、CDK1(PDB ID:4Y72)、IL6(PDB ID:1ALU)、CXCR4(PDB ID:3ODU)进行分子对接,结果显示,党参炔苷、黄豆黄素、紫丁香苷、党参碱和党参苷Ⅰ与核心靶点间具有较高的结合能,结合能均小于-5 kcal/mol,表明具有较好的结合能力[18],见表 2和图 11(颜色越深表示结合越好),借助DS软件对结果进行可视化,见图 12。

|
|
| 图 11 活性成分与靶点对接结果热图 Fig. 11 Heat map of active ingredient-target docking results |

|
| 图 12 活性成分与靶点对接模式图 Fig. 12 Diagram of active ingredient-target docking patterns |
生物信息学和网络药理学方法为系统性明确药物-靶点-疾病网络提供了可靠的模型,同时运用机器学习的方法,将多维数据进行整合,可实现对数据的分析和筛选,更加明确靶点的筛选范围[19],有助于从整体上理解中药多成分、多靶点、多通路的协同作用。网络药理学和机器学习技术相结合,为中药防治疾病提供了一种新思路与方法。
本研究结果显示,HIF-1和PI3K-Akt信号通路可能是党参改善低氧适应的关键通路。目前大量研究已证实,HIF-1信号通路在多种生理和病理过程中发挥重要作用,是细胞适应缺氧环境的关键机制。而PI3K-Akt信号通路可通过介导HIF-1A的活性调控HIF-1信号通路亦发挥调节缺氧的作用[20]。此外,通过研究笔者筛选得到了党参改善低氧适应的7个核心靶点,其中HIF1A、CDK1、KDR、IL-6和CXCR4等靶点与HIF-1信号通路密切相关,均为机体低氧适应的关键靶点。尤其是HIF1A,其活化以及介导的下游途径是低氧状态下细胞增殖和机体适应缺氧环境的重要机制。在缺氧条件下,HIF1A蛋白被稳定,HIF1A的表达可促进细胞对缺氧环境的适应和存活[21-22],同时还可调节多种线粒体呼吸链酶的表达,从而改善细胞代谢和能量利用[23]。CDK1是细胞周期调控中的一个关键酶,主要负责细胞从G2期进入M期的调控,在细胞周期中发挥核心作用,同时在转录调控、细胞增殖与适应缺氧等过程中也具有重要功能。Liu等[24]研究发现,缺氧会诱导脂质积累,导致线粒体功能障碍,使CDK1表达减少。由于缺氧会导致HIF1A的稳定与积累,HIF1A可以上调CDK抑制蛋白p21,从而抑制CDK1活性,提高细胞的低氧适应能力[25]。KDR是血管内皮生长因子(VEGF)的特异性受体,当缺氧发生时,HIF1A的活化可以激活下游VEGF基因的表达,当VEGF与KDR结合时,KDR的酪氨酸激酶活性被激活,可触发下游PI3K/Akt信号通路,从而增强对缺氧环境的适应能力[26]。目前KDR作为血管生成的关键调控因子,在多种缺血、缺氧性疾病中的重要作用已受到广泛研究,因此通过靶向KDR来缓解低氧有一定的应用前景。此外,低氧会诱发机体产生一定的炎症反应,而IL-6在低氧环境下可以通过自分泌或旁分泌作用,影响周围细胞的功能,激活HIF-1信号通路,促进细胞存活和适应缺氧环境[27]。CXCR4是一种G蛋白偶联受体,同样为HIF-1α的下游靶点,研究表明CXCR4与低氧微环境密切相关,低氧可以直接诱导内皮细胞和造血干细胞中CXCR4表达增加,CXCR4通过与其配体CXCL12结合,激活下游PI3K/Akt信号通路,可以增强细胞的低氧适应能力,使细胞更好地应对低氧环境[28]。
基于机器学习结合网络药理学的方法,本研究明确了党参炔苷、黄豆黄素、紫丁香苷、党参碱和党参苷Ⅰ等可能是党参改善人体低氧适应的潜在有效成分。目前已证实,党参炔苷具有抗氧化、抗缺氧药理活性[29]。Wang等[30]研究表明党参炔苷可以保护小胶质细胞(BV2)免受缺氧诱导的损伤,并通过降低缺氧下BV2细胞中HIF-1α和IL-6的表达来减轻缺氧损伤。Ni等[31]实验证明,党参炔苷可以改善小鼠贫血模型中局部组织氧供应不足的情况,显示出较好的抗缺氧作用。研究表明,黄豆黄素具有较好的抗氧化活性[32-33]。Dong等[34]实验显示,黄豆黄素通过降低鱼藤酮诱导的氧化应激而表现出神经保护作用;可以减少缺氧所导致的HIF-1α积累[35]。此外,黄豆黄素还具有调节CXCR4的作用,展现出一定的抗缺氧作用潜力[36]。紫丁香苷具有很好的药理活性,可有效缓解缺氧所导致的脑损伤,其作用机制主要聚焦于HIF-1和PI3K/Akt信号通路[37],证实了其通过调控HIF-1信号通路发挥治疗作用的可行性。Zhou等[38]的网络药理学分析同样显示紫丁香苷的药理作用途径聚焦在HIF-1信号通路上,并结合细胞实验证实了紫丁香苷能提高缺氧下海马神经元细胞的存活率,表明其具有较好的抗缺氧活性。目前研究表明,党参苷Ⅰ可能主要通过增加超氧化物歧化酶(SOD)和谷胱甘肽过氧化物酶(GSH-Px)的活性来抵抗缺氧所导致的氧化应激,并通过抑制IL-6的表达发挥一定的抗缺氧作用[39-41]。综上,党参炔苷、黄豆黄素、紫丁香苷和党参苷Ⅰ等党参活性成分具有一定的抗缺氧作用,基于现有文献报道也验证了该研究预测结果的可靠性。
综上所述,该研究采用机器学习结合网络药理学的方法,系统探讨了“补气良药”党参提高人体低氧适应能力的潜在药效成分及分子机制。结果表明党参炔苷、黄豆黄素、紫丁香苷、党参苷Ⅰ和党参碱可能是党参提高人体低氧适应的潜在有效成分。进一步明确了HIF-1信号通路在党参提高人体低氧适应能力中起主导作用,HIF1A、KDR和CKD1等可能是其关键作用靶点。笔者研究明确了党参改善机体低氧适应的临床治疗潜力,为后续基础研究以及临床应用提供了参考和思路,亦有助于阐释党参“补气”功效的科学内涵。
| [1] |
赵丽, 李杰. 生命的低氧适应: 2019年度诺贝尔生理学或医学奖成果解析[J]. 科技导报, 2020, 38(2): 79-85. |
| [2] |
沈安然, 王彬, 刘必成. 氧感受与适应机制的研究进展[J]. 生理学报, 2020, 72(5): 660-666. |
| [3] |
LEE P, CHANDEL N S, SIMON M C. Cellular adaptation to hypoxia through hypoxia inducible factors and beyond[J]. Nature Reviews Molecular Cell Biology, 2020, 21(5): 268-283. DOI:10.1038/s41580-020-0227-y |
| [4] |
LUO Z, TIAN M F, YANG G, et al. Hypoxia signaling in human health and diseases: Implications and prospects for therapeutics[J]. Signal Transduction and Targeted Therapy, 2022, 7(1): 218. DOI:10.1038/s41392-022-01080-1 |
| [5] |
国家药典委员会. 中华人民共和国药典-一部: 2020年版[S]. 北京: 中国医药科技出版社, 2020.
|
| [6] |
DONG J J, NA X, HOU A J, et al. A review of the botany, ethnopharmacology, phytochemistry, analysis method and quality control, processing methods, pharmacological effects, pharmacokinetics and toxicity of Codonopsis Radix[J]. Frontiers in Pharmacology, 2023, 14: 1162036. DOI:10.3389/fphar.2023.1162036 |
| [7] |
GAO S M, LIU J S, WANG M, et al. Traditional uses, phytochemistry, pharmacology and toxicology of Codonopsis: A review[J]. Journal of Ethnopharmacology, 2018, 219: 50-70. DOI:10.1016/j.jep.2018.02.039 |
| [8] |
牛明, 张斯琴, 张博, 等. 《网络药理学评价方法指南》解读[J]. 中草药, 2021, 52(14): 4119-4129. |
| [9] |
RU J L, LI P, WANG J N, et al. TCMSP: A database of systems pharmacology for drug discovery from herbal medicines[J]. Journal of Cheminformatics, 2014, 6: 13. DOI:10.1186/1758-2946-6-13 |
| [10] |
LIU Z Y, GUO F F, WANG Y, et al. BATMAN-TCM: A bioinformatics analysis tool for molecular mechanism of traditional Chinese medicine[J]. Scientific Reports, 2016, 6: 21146. DOI:10.1038/srep21146 |
| [11] |
WANG Z H, LIANG L, YIN Z, et al. Improving chemical similarity ensemble approach in target prediction[J]. Journal of Cheminformatics, 2016, 8: 20. DOI:10.1186/s13321-016-0130-x |
| [12] |
NICKEL J, GOHLKE B O, EREHMAN J, et al. SuperPred: Update on drug classification and target prediction[J]. Nucleic Acids Research, 2014, 42: W26-W31. DOI:10.1093/nar/gku477 |
| [13] |
WANG Q, QIAO W Y, ZHANG H H, et al. Nomogram established on account of Lasso-Cox regression for predicting recurrence in patients with early-stage hepatocellular carcinoma[J]. Frontiers in Immunology, 2022, 13: 1019638. DOI:10.3389/fimmu.2022.1019638 |
| [14] |
MALTAROLLO V G, KRONENBERGER T, ESPINOZA G Z, et al. Advances with support vector machines for novel drug discovery[J]. Expert Opinion on Drug Discovery, 2019, 14(1): 23-33. DOI:10.1080/17460441.2019.1549033 |
| [15] |
LIU Z X, WANG P Q, ZHANG Q F, et al. iHypoxia: An integrative database of protein expression dynamics in response to hypoxia in animals[J]. Genomics, Proteomics & Bioinformatics, 2023, 21(2): 267-277. |
| [16] |
YUE J Q, XIAO Y, CHEN W S. Insights into genus Codonopsis: From past achievements to future perspectives[J]. Critical Reviews in Analytical Chemistry, 2024, 54(8): 3345-3376. DOI:10.1080/10408347.2023.2242953 |
| [17] |
兰晓燕, 周利, 李翔, 等. 党参的研究进展及其质量标志物的预测分析[J]. 中国中药杂志, 2023, 48(8): 2020-2040. |
| [18] |
HSIN K Y, GHOSH S, KITANO H. Combining machine learning systems and multiple docking simulation packages to improve docking prediction reliability for network pharmacology[J]. PLoS One, 2013, 8(12): e83922. DOI:10.1371/journal.pone.0083922 |
| [19] |
段贤春, 黄石, 彭代银, 等. 网络药理学在中药复方研究中的应用[J]. 中国药理学通报, 2020, 36(3): 303-308. |
| [20] |
DONG S H, LIANG S, CHENG Z Q, et al. ROS/PI3K/Akt and Wnt/β-catenin signalings activate HIF-1α-induced metabolic reprogramming to impart 5-fluorouracil resistance in colorectal cancer[J]. Journal of Experimental & Clinical Cancer Research, 2022, 41(1): 15. |
| [21] |
WEI X Y, ZHAO L, REN R Z, et al. MiR-125b loss activated HIF1α/pAKT loop, leading to transarterial chemoembolization resistance in hepatocellular carcinoma[J]. Hepatology, 2021, 73(4): 1381-1398. DOI:10.1002/hep.31448 |
| [22] |
KOEDDERITZSCH K, ZEZINA E, LI L Z, et al. TNF induces glycolytic shift in fibroblast like synoviocytes via GLUT1 and HIF1A[J]. Scientific Reports, 2021, 11(1): 19385. DOI:10.1038/s41598-021-98651-z |
| [23] |
GERMANOVA E, KHMIL N, PAVLIK L, et al. The role of mitochondrial enzymes, succinate-coupled signaling pathways and mitochondrial ultrastructure in the formation of urgent adaptation to acute hypoxia in the myocardium[J]. International Journal of Molecular, 2022, 23(22): 14248. |
| [24] |
LIU L M, LIU T, JIA R, et al. Downregulation of fatty acid oxidation led by Hilpda increases G2/M arrest/delay-induced kidney fibrosis[J]. Biochimica et Biophysica Acta(BBA)-Molecular Basis of Disease, 2023, 1869(5): 166701. DOI:10.1016/j.bbadis.2023.166701 |
| [25] |
ZHAO S, ZHOU L L, DICKER D T, et al. Anti-cancer efficacy including Rb-deficient tumors and VHL-independent HIF1α proteasomal destabilization by dual targeting of CDK1 or CDK4/6 and HSP90[J]. Scientific Reports, 2021, 11(1): 20871. DOI:10.1038/s41598-021-00150-8 |
| [26] |
SINGH N K, HANSEN D E 3rd, KUNDUMANI-SRIDHARAN V, et al. Both Kdr and Flt1 play a vital role in hypoxia-induced Src-PLD1-PKCγ-cPLA(2) activation and retinal neovascularization[J]. Blood, 2013, 121(10): 1911-1923. DOI:10.1182/blood-2012-03-419234 |
| [27] |
XUE H, YUAN G, GUO X, et al. A novel tumor-promoting mechanism of IL6 and the therapeutic efficacy of tocilizumab: Hypoxia-induced IL6 is a potent autophagy initiator in glioblastoma via the p-STAT3-MIR155-3p-CREBRF pathway[J]. Autophagy, 2016, 12(7): 1129-1152. DOI:10.1080/15548627.2016.1178446 |
| [28] |
GUO F, WANG Y, LIU J, et al. CXCL12/CXCR4:A symbiotic bridge linking cancer cells and their stromal neighbors in oncogenic communication networks[J]. Oncogene, 2016, 35(7): 816-826. DOI:10.1038/onc.2015.139 |
| [29] |
YOON I S, CHO S S. Effects of lobetyolin on xanthine oxidase activity in vitro and in vivo: Weak and mixed inhibition[J]. Natural Product Research, 2021, 35(10): 1667-1670. DOI:10.1080/14786419.2019.1622108 |
| [30] |
WANG J, LIU X, WEI W Y, et al. Regulation of oxygen-glucose deprivation/reperfusion-induced inflammatory responses and M1-M2 phenotype switch of BV2 microglia by lobetyolin[J]. Metabolic Brain Disease, 2023, 38(8): 2627-2644. DOI:10.1007/s11011-023-01292-6 |
| [31] |
NI S H, ZHANG X J, OUYANG X L, et al. Lobetyolin alleviates ferroptosis of skeletal muscle in 5/6 nephrectomized mice via activation of hedgehog-GLI1 signaling[J]. Phytomedicine, 2023, 115: 154807. DOI:10.1016/j.phymed.2023.154807 |
| [32] |
王艳梅, 吕立勋. 黄豆苷元的药理作用及临床应用[J]. 中国药师, 2007, 10(9): 910-912. |
| [33] |
张家瑞, 孙达亮, 石娟娟, 等. 大豆黄素抗抑郁作用及其机制研究[J]. 中国新药杂志, 2015, 24(13): 1531-153. |
| [34] |
DONG N, YANG Z L. Glycitein exerts neuroprotective effects in Rotenone-triggered oxidative stress and apoptotic cell death in the cellular model of Parkinson's disease[J]. Acta Biochimica Polonica, 2022, 69(2): 447-452. |
| [35] |
SINGH-GUPTA V, ZHANG H, YUNKER C K, et al. Daidzein effect on hormone refractory prostate cancer in vitro and in vivo compared to genistein and soy extract: Potentiation of radiotherapy[J]. Pharmaceutical Research, 2010, 27(6): 1115-1127. DOI:10.1007/s11095-010-0107-9 |
| [36] |
VAN DER EECKEN H, JONIAU S, BERGHEN C, et al. The use of soy isoflavones in the treatment of prostate cancer: A focus on the cellular effects[J]. Nutrients, 2023, 15(23): 4856. DOI:10.3390/nu15234856 |
| [37] |
ZHOU Y Y, CHENG C L, BARANENKO D, et al. Effects of Acanthopanax senticosus on brain injury induced by simulated spatial radiation in mouse model based on pharmacokinetics and comparative proteomics[J]. International Journal of Molecular Sciences, 2018, 19(1): 159. DOI:10.3390/ijms19010159 |
| [38] |
ZHOU F, HE K, GUAN Y, et al. Network pharmacology-based strategy to investigate pharmacological mechanisms of Tinospora sinensis for treatment of Alzheimer's disease[J]. Journal of Ethnopharmacology, 2020, 259: 112940. DOI:10.1016/j.jep.2020.112940 |
| [39] |
XIE Q, HU X R, ZHAO X, et al. Effects and mechanism of extracts rich in phenylpropanoids-polyacetylenes and polysaccharides from Codonopsis Radix on improving scopolamine-induced memory impairment of mice[J]. Journal of Ethnopharmacology, 2024, 319(Pt 1): 117106. |
| [40] |
CHEN X Y, SHAO R, WANG Y. Multi-layered effects of Codonopsis Radix on the immune system[J]. Acupuncture and Herbal Medicine, 2024, 4(3): 405-419. DOI:10.1097/HM9.0000000000000130 |
| [41] |
CHEN Z H, SU Y X, DING J T, et al. Lobetyolin protects mice against LPS-induced sepsis by downregulating the production of inflammatory cytokines in macrophage[J]. Frontiers in Pharmacology, 2024, 15: 1405163. DOI:10.3389/fphar.2024.1405163 |
2. State Key Laboratory of Component-Based Chinese Medicine, Tianjin 301617, China;
3. National Key Laboratory of Chinese Medicine Modernization, Tianjin 301617, China;
4. Key Laboratory of Pharmacology of Traditional Chinese Medical Formulae, Ministry of Education, Tianjin 301617, China
 2025, Vol. 42
2025, Vol. 42