文章信息
- 刘杰, 田晓轩, 崔英, 朱彦
- LIU Jie, TIAN Xiao-xuan, CUI Ying, ZHU Yan
- 药用动物乌梢蛇线粒体基因组全序列分析
- Analysis of mitochondrial genome of Ptyas dhumnades
- 天津中药大学学报, 2016, 35(3): 187-191
- Journal of Tianjin University of Traditional Chinese Medicine, 2016, 35(3): 187-191
- http://dx.doi.org/10.11656/j.issn.1673-9043.2016.03.10
-
文章历史
收稿日期: 2016-02-24
乌梢蛇Ptyas dhumnades(Cantor,1842)在分类上属于蛇亚目(Serpentes)游蛇科(Colubridae)游蛇亚科(Colubrinae)鼠蛇属(Ptyas)。历史上,其异名包括Zaocys dhumnad(Cope,1860),Ablabes vittatus(Duméril,Bibron & Duméril,1854) 及Coluber dhumnades(Cantor,1842) 等,后被归入Ptyas属[1-2]。本物种俗名中国鼠蛇(Chinese rat snake)、大眼鼠蛇(Big-eyed rat snake),或俗称过山刀。该物种主要分布于中国,生存于海拔50~1 570 m的区域,在河北、河南、山西、陕西、甘肃、四川、湖北、湖南、江西、江苏、福建、云南、贵州、广东、广西、台湾等省份均有发现[3]。乌梢蛇属无毒蛇类,其分类除去内脏的干燥体,有祛风、通络、止痉的功效,常用于治疗风湿顽痹、抽搐痉挛、瘰疠恶疮等症[4]。其分类特征包括头部鳞呈黑褐色,脊背耸立成屋脊状,背鳞有端窝两个,行数为偶数等[5-6]。近年来,基于CYTB及CO1基因序列以及高特异性聚合酶链式反应(PCR)的乌梢蛇分子鉴定方法也得到建立与应用[7-9]。
动物线粒体基因组包含了从分子序列到基因结构的全面信息,其基因组成排列相对保守,具母系遗传特征,且易于扩增测序[10],被广泛用于动物系统发育,以及种群保护等领域研究。蛇类的线粒体基因组编码2个rRNA、22个左右的tRNA和13个蛋白,1~2个非编码的控制区,并常包括1个轻链复制起始区。本文采用Ion torrent PGM二代测序技术[11],获得了乌梢蛇线粒体DNA全序列,此为国内外有关鼠蛇属动物线粒体基因组的首次公开报道。
1 材料与方法 1.1 动物乌梢蛇购于福建福州,并经形态学及CO1基因分子鉴定。取新鲜肌肉组织立即使用。Voucher ID为wss-1。
1.2 总DNA提取参照经典的酚氯仿抽提方法,获得总基因组DNA,并经A260/A280粗略估计浓度及纯度。
1.3 Ion Torrent PGM高通量测序 1.3.1 鸟枪法建库鸟枪法是指将目的DNA随机打断成大小不同的片段,再将这些片段的序列连接起来的测序方法。
1.3.2 OneTouch(油包水PCR)每个微小磁珠通过接头连接相应的DNA片段,然后把含有DNA片段的磁珠和扩增试剂的溶液注入矿物油中,形成了油包水的小体系,使得每个小水滴只含一个磁珠,每个小水滴就是一个微反应体系。通过扩增,DNA片段可以增加几百万个拷贝,并且都连接在磁珠上。
1.3.3 ES富集油包水PCR之后,加入Dynabeads® MyOneTM Streptavidn C1 beads,它通过亲和素-生物素与带有双链模板的磁珠结合,最后加入氢氧化钠(NaOH)使得双链解链,制备单链模板。
1.3.4 PGM上机测序将带有单链模板的磁珠、测序引物、DNA聚合酶混匀,离心使其进入318芯片的微孔中,然后上机测序。
1.4 序列拼接从测序服务器上下载鸟枪法文库测序的原始数据至本地,预处理过滤掉低质量序列、过短序列以及错误序列,根据目的基因组大小(约16K bp),分割文件,调整合适的覆盖倍数(基因组大小的20X为佳),使用MIRA 4.0进行de novo拼接,结合近缘物种mapping组装,经反复迭代补全控制区(重复区)后,进行最高质量碱基合意,得到合意后的完整线粒体全基因组序列。
1.5 注释分析和提交使用MITOS server线粒体在线注释系统[12],对全序列进行注释,结合tRNAscan-SE 1.21软件,进行tRNA定位。以游蛇亚科Elaphe anomala,Elaphe bimaculata,Oligodon ningshaanensis和Euprepiophis perlacea的线粒体基因组全序列为参照,对蛋白质编码基因、tRNA、rRNA及控制区等进行注释,L链复制起始区二级结构由RNA structure折叠生成。使用软件MEGA 4.0及Bioedit等统计序列总长、碱基组成和GC含量等。完整的乌梢蛇线粒体基因组全序列经Sequin注释后,提交GenBank,登录号为KR812112。
2 结果 2.1 序列拼接结果获取乌梢蛇线粒体基因组后,经bowtie2比对[13],确认共189 977条序列(reads)可拼接至该基因组,序列平均长度214.4 bp,测序深度达1 958倍。
2.2 整体结构乌梢蛇线粒体基因组结构见图 1、表 1。与大多数脊椎动物类似,乌梢蛇共编码37个基因,包括了22个tRNA,13个蛋白编码基因,2个rRNA(16S rRNA、12S rRNA)并有1个轻链(L链)复制起始区。其中除ND6和8个tRNA(Ala、Asn、Cys、Gln、Glu、Pro、Ser和Tyr)由L链编码,其余28个基因均由重链(H链)编码。其基因的排布顺序与大部分游蛇类动物相同。
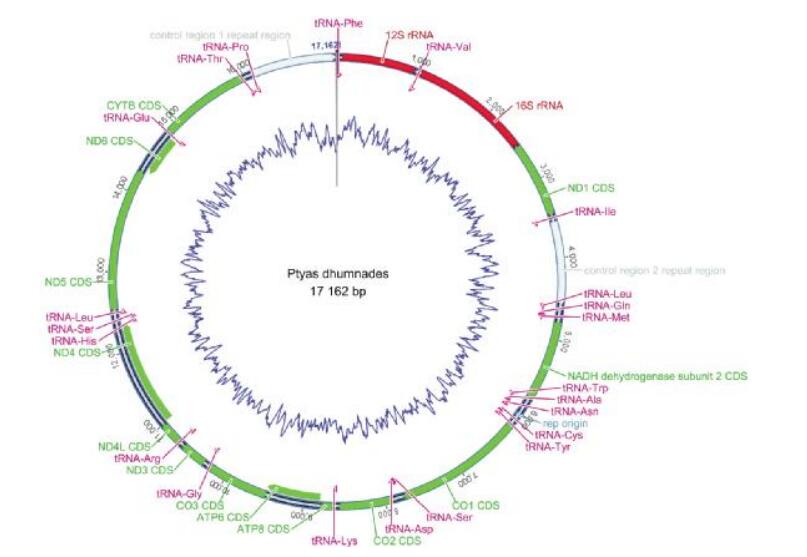
|
| 图 1 乌梢蛇线粒体基因组结构图 ND 1~6/4l:NADH dehydrogenase subunit 1~6/4l;ATP6/8:ATP synthase F0 subunit 6/8;CO 1-3:cytochrome c oxidase subunit I-III;CYTB:cytochrome b;箭头方向表示基因的转录方向;曲线示各个位点GC含量。 |
| 名称 | 链 | 起始位置 | 终止位置 | 长度 (bp) |
反密码子 | 起始密码子 | 终止密码子 | 间隔( + ) /重叠(-) |
| tRNA-Phe | H | 1 | 61 | 61 | GAA | |||
| 12S rRNA | H | 62 | 982 | 921 | ||||
| tRNA-Val | H | 983 | 1 045 | 63 | TAC | |||
| 16S rRNA | H | 1 046 | 2 526 | 1 481 | ||||
| ND1CDS | H | 2 527 | 3 490 | 964 | ATA | T-- | ||
| tRNA-Ile | H | 3 491 | 3 558 | 68 | GAT | |||
| control region 2 | H | 3 559 | 4 580 | 1 022 | ||||
| tRNA-Leu | H | 4581 | 4 653 | 73 | TAA | |||
| tRNA-Gln | L | 4 655 | 4 725 | 71 | TTG | +1 | ||
| tRNA-Met | H | 4 727 | 4 790 | 64 | CAT | +1 | ||
| ND2 CDS | H | 4 791 | 5 820 | 1 030 | ATT | T-- | ||
| tRNA-Trp | H | 5 821 | 5 885 | 65 | TCA | |||
| tRNA-Ala | L | 5 886 | 5 950 | 65 | TGC | |||
| tRNA-Asn | L | 5 951 | 6 022 | 72 | GTT | |||
| rep origin | H | 6 023 | 6 055 | 33 | ||||
| tRNA-Cys | L | 6 056 | 6115 | 60 | GCA | |||
| tRNA-Tyr | L | 6116 | 6 178 | 63 | GTA | |||
| CO1CDS | H | 6 180 | 7 781 | 1 602 | GTG | AGA | +1 | |
| tRNA-Ser | L | 7 772 | 7 838 | 67 | TGA | -10 | ||
| tRNA-Asp | H | 7 839 | 7 901 | 63 | GTC | |||
| CO2CDS | H | 7 903 | 8 587 | 685 | ATG | T-- | +1 | |
| tRNA-Lys | H | 8 588 | 8 650 | 63 | TTT | |||
| ATP8CDS | H | 8 652 | 8 810 | 159 | ATG | TAA | +1 | |
| ATP6CDS | H | 8 801 | 9 481 | 681 | ATG | TAA | -10 | |
| CO3CDS | H | 9 481 | 10 264 | 784 | ATG | T-- | -1 | |
| tRNA-Gly | H | 10 265 | 10 325 | 61 | TCC | |||
| ND3CDS | H | 10 326 | 10 668 | 343 | ATT | T-- | ||
| tRNA-Arg | H | 10 669 | 10 732 | 64 | TCG | |||
| ND4LCDS | H | 10 733 | 11 023 | 291 | ATG | TAA | ||
| ND4CDS | H | 11 023 | 12 359 | 1 337 | ATG | TA- | -1 | |
| tRNA-His | H | 12 360 | 12 424 | 65 | GTG | |||
| tRNA-Ser | H | 12 425 | 12481 | 57 | GCT | |||
| tRNA-Leu | H | 12 479 | 12 549 | 71 | TAG | -3 | ||
| ND5CDS | H | 12 550 | 14316 | 1 767 | ATG | TAG | ||
| ND6CDS | L | 14313 | 14819 | 507 | ATG | TAG | -4 | |
| tRNA-Glu | L | 14 829 | 14 890 | 62 | TTC | +9 | ||
| CYTBCDS | H | 14 891 | 16 007 | 1 117 | ATG | T-- | ||
| tRNA-Thr | H | 16 008 | 16 075 | 68 | TGT | |||
| tRNA-Pro | L | 16 076 | 16 137 | 62 | TGG | |||
| control region 1 | H | 16 138 | 17 162 | 1 025 |
乌梢蛇线粒体基因组全长17 162 bp,其中L链的碱基含量为:A=5 932 bp(34.6%),C=4 843 bp(28.2%),G=2 139 bp(12.5%),T=4 248 bp(24.8%),GC=6 982 bp(40.7%),A > C > T >G,AT% > GC%。且呈AT偏斜(0.165)和CG偏斜(0.387),这也与蛇类普遍情况相同。
2.4 蛋白编码基因乌梢蛇线粒体基因组中蛋白编码基因全长11 267 bp,约占全基因组的65.65%。A=3 967 bp(35.2%),C=3 298 bp(29.3%),G=1 267 bp(11.2%),T=2 735 bp(24.3%),GC=4 565 bp(40.5%),A > C > T >G,AT% > GC%。所有蛋白编码基因无内含子。除ND1以ATA为起始密码子,ND2和ND3以ATT起始,CO1以GTG起始外,其余均使用ATG为起始密码子。除CO1以AGA为终止密码子,ND5和ND6以TAG终止,其余蛋白均使用T或TA碱基终止,并通过转录过程中3’端的poly A补全,形成完整终止密码子。氨基酸使用频率的分析见表 2。在一共3 747个氨基酸中,出现频率较低的为Cys(0.77%)、Asp(1.52%)和Arg(1.65%),而频率较高的为Leu(14.87%)和Thr(12.09%)。
| 基因及氨基酸名称 | Ala | Cys | Asp | Glu | Phe | Gly | His | Ile | Lys | Leu | Met | Asn | Pro | Gln | Arg | Ser | Thr | Val | Trp | Tyr |
| ND1 | 21 | 1 | 3 | 11 | 14 | 17 | 3 | 19 | 8 | 61 | 22 | 15 | 21 | 8 | 7 | 25 | 39 | 6 | 8 | 12 |
| ND2 | 24 | 1 | 2 | 5 | 7 | 9 | 7 | 36 | 11 | 49 | 29 | 16 | 19 | 5 | 4 | 30 | 67 | 5 | 10 | 7 |
| CO1 | 36 | 4 | 13 | 9 | 45 | 45 | 18 | 46 | 12 | 62 | 34 | 19 | 32 | 7 | 9 | 35 | 42 | 30 | 19 | 16 |
| CO2 | 16 | 3 | 8 | 17 | 7 | 10 | 9 | 14 | 3 | 27 | 12 | 6 | 13 | 12 | 6 | 14 | 23 | 15 | 5 | 8 |
| ATP8 | 1 | 0 | 1 | 0 | 1 | 0 | 2 | 3 | 3 | 9 | 5 | 1 | 6 | 2 | 1 | 0 | 11 | 1 | 3 | 2 |
| ATP6 | 13 | 2 | 1 | 6 | 9 | 8 | 4 | 19 | 5 | 42 | 21 | 12 | 14 | 6 | 3 | 15 | 28 | 11 | 3 | 4 |
| CO3 | 14 | 1 | 5 | 9 | 19 | 19 | 16 | 15 | 7 | 34 | 11 | 3 | 11 | 6 | 4 | 16 | 30 | 18 | 12 | 11 |
| ND3 | 5 | 1 | 3 | 5 | 5 | 5 | 1 | 17 | 2 | 21 | 5 | 6 | 8 | 2 | 1 | 3 | 16 | 2 | 3 | 3 |
| ND4L | 7 | 2 | 1 | 2 | 4 | 3 | 3 | 9 | 3 | 18 | 8 | 2 | 2 | 2 | 1 | 7 | 12 | 9 | 0 | 1 |
| ND4 | 31 | 4 | 3 | 10 | 20 | 15 | 11 | 36 | 12 | 82 | 41 | 14 | 24 | 15 | 9 | 30 | 58 | 8 | 9 | 13 |
| ND5 | 36 | 3 | 9 | 12 | 23 | 25 | 18 | 61 | 24 | 81 | 50 | 37 | 26 | 17 | 8 | 43 | 84 | 11 | 10 | 10 |
| ND6 | 5 | 5 | 2 | 3 | 14 | 18 | 0 | 10 | 3 | 28 | 6 | 3 | 2 | 2 | 3 | 12 | 4 | 35 | 5 | 8 |
| CYTB | 22 | 2 | 6 | 5 | 30 | 20 | 14 | 37 | 9 | 43 | 20 | 21 | 21 | 7 | 6 | 29 | 39 | 17 | 12 | 12 |
| sum | 231 | 29 | 57 | 94 | 198 | 194 | 106 | 322 | 102 | 557 | 264 | 155 | 199 | 91 | 62 | 259 | 453 | 168 | 99 | 107 |
| % | 6.16 | 0.77 | 1.52 | 2.51 | 5.28 | 5.18 | 2.83 | 8.59 | 2.72 | 14.87 | 7.05 | 4.14 | 5.31 | 2.43 | 1.65 | 6.91 | 12.09 | 4.48 | 2.64 | 2.86 |
乌梢蛇的12S rRNA和16S rRNA长度分别为921 bp和1 481 bp,GC含量分别为44.7%和40.2%。22个tRNA基因中,最短的为tRNA-Ser(anticodon=GCT,57 bp),最长的为tRNA-Leu(anticodon=TAA,73 bp)。大部分tRNA可折叠成三叶草二级结构,但tRNA-Ser(anticodon=GCT)折叠后缺失D臂,tRNA-Cys缺失TΨC环,见图 2。

|
| 图 2 tRNA-Ser(anticodon=GCT),tRNA-Cys及L链复制起始区二级结构图 A:tRNA-Ser(anticodon=GCT);B:tRNA-Cys;C:L链复制起始区 |
乌梢蛇的1号控制区(control region 1)位于tRNA-Pro与tRNA-Phe之间,2号控制区(control region 2)位于tRNA-Ile与tRNA-Leu(anticodon=TAA)之间,长度分别为1 025 bp和1 022 bp,比对后发现两者一致碱基为1 018 bp,一致率为99.5%。除控制区外,乌梢蛇线粒体基因组中有14 bp的6个非编码间隔区域,其中最长的间隔区为9 bp,位于ND6和tRNA-Glu之间,显示该物种线粒体基因组结构较为紧凑。
2.7 L链复制起始区乌梢蛇基因组的L链复制起始区,长度为33 bp,可折叠成稳定的茎环结构,见于图 2C。其中茎由9对碱基组成,环为11个碱基。
3 讨论线粒体全基因组测序方法,在以往多基于PCR技术[14],该方法需根据目标物种的近缘物种,设计合适引物,进行普通PCR或Long-PCR( > 4 kb),并对扩增产物进行测序或步移测序。而近年来,随着高通量测序技术的成熟,测序成本的大幅下降,更多的目光集中在利用总基因组DNA测序结果,从中筛选拼接线粒体基因组序列[15-17]。本实验即采取后者策略,建立了基于高通量测序结果的线粒体基因组拼接工作流程。此方法可快速、低成本的得到目标物种的高深度线粒体基因组,并避免PCR过程中产生的碱基错配可能。
在系统发育学研究中,线粒体中蛋白编码区以及tRNA区的结构变化应用较多。而另一方面,控制区(重复区)序列由于受进化压力较小,其碱基替换速率较快,这对药用动物的种下研究尤有意义。与其他真蛇类动物类似,本研究同样在结构紧凑的线粒体基因组中,发现了两个控制区序列,其形成原因尚无定论。但已有研究发现,蛇类中这两个控制区在同一物种内相似度很高,而不同的物种间又有较大的分歧。这提示了其可能是协同进化的结果[18]。
中药资源的研究是目前中药研究的热点问题,较多的工作集中在药用植物上[19-20],而动物上开展较少。通过线粒体基因组数据的累积,研究以上双控制区序列在种内(种群间)的变化,也可作为研究药用蛇类种群分化的分子标记,为药用蛇类的资源普查及保护提供参考。
| [1] | David P, Vogel G. The Snakes of Sumatra:an annotated checklist and key with natural history notes[M]. Germany:Frankfurt am Main:Chimaira, 1996. |
| [2] | David P, Das I. On the grammar of the gender of Ptyas Fitzinger, 1843(Serpentes:Colubridae)[J]. Hamaddryad, 2004, 28 (1-2) : 113–116. |
| [3] | 李军德, 黄璐琦, 曲晓波. 中国药用动物志[M]. 福州: 福建科学技术出版社, 2013 |
| [4] | 国家药典委员会. 中华人民共和国药典(一部)[S]. 北京:中国医药科技出版社, 2010. |
| [5] | 宋秀菊. 动物乌梢蛇的鉴别[J]. 时珍国医国药, 2012, 23 (7) : 1824. |
| [6] | 林喆, 胡丽娜, 李娜. 动物药整理研究——乌梢蛇[J]. 吉林中医药, 2009, 29 (11) : 982–984. |
| [7] | 廖婧, 梁镇标, 张亮, 等. 常见药用蛇类的DNA条形码研究[J]. 中国药学杂志, 2013, 48 (15) : 1255–1260. |
| [8] | 唐晓晶, 冯成强, 黄璐琦, 等. 高特异性PCR方法鉴别乌梢蛇及其混淆品[J]. 中国药学杂志, 2007, 42 (5) : 333–336. |
| [9] | 王义权, 周开亚, 徐珞珊, 等. 中药材乌梢蛇及其混淆品的DNA序列分析鉴别[J]. 药学学报, 1999, 34 (1) : 68–72. |
| [10] | Wolstenholme DR. Animal mitochondrial DNA:structure and evolution[J]. Int Rev Cytol, 1992, 141 (6) : 173–216. |
| [11] | Morinha F, Clemente C, Cabral JA, et al. Next-generation sequencing and comparative analysis of Pyrrhocorax pyrrhocorax and Pyrrhocorax graculus (Passeriformes:Corvidae) mitochondrial genomes[J]. Mitochondrial DNA, 2014 (28) : 1–4. |
| [12] | Bernt M, Donath A, Juhling F, et al. MITOS:improved denovo metazoan mitochondrial genome annotation[J]. Mol Phylogenet Evol, 2013, 69 (2) : 313–319. |
| [13] | Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9 (4) : 357–359. |
| [14] | Zardoya R, Suárez M. Sequencing and phylogenomic analysis of whole mitochondrial genomes of animals[J]. Methods in Molecular Biology, 2007, 422 : 185–200. |
| [15] | Tang M, Tan M, Meng G, et al. Multiplex sequencing of pooled mitochondrial genomes-a crucial step toward biodiversity analysis using mito-metagenomics[J]. Nucleic Acids Res, 2014, 42 (22) : e166. |
| [16] | Gillett CP, Crampton-Platt A, Timmermans MJ, et al. Bulk de novo mitogenome assembly from pooled total DNA elucidates the phylogeny of weevils (Coleoptera:Curculionoidea)[J]. Mol Biol Evol, 2014, 31 (8) : 2223–2237. |
| [17] | Hahn C, Bachmann L, Chevreux B. Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads——a baiting and iterative mapping approach[J]. Nucleic Acids Res, 2013, 41 (13) : e129. |
| [18] | Jiang ZJ, Castoe TA, Austin CC, et al. Comparative mitochondrial genomics of snakes:extraordinary substitution rate dynamics and functionality of the duplicate control region[J]. BMC Evol Biol, 2007 (7) : 123. |
| [19] | 李天祥, 李国辉, 张春艳, 等. 天津黄精属植物的资源考察[J]. 天津中医药, 2013, 30 (12) : 749–753. |
| [20] | 田春雨, 薄海美, 许静, 等. 唐山山区野生药用植物资源及其分布组成特点的初步研究[J]. 天津中医药, 2014, 31 (1) : 56–58. |
 2016, Vol. 35
2016, Vol. 35




