文章信息
- 杨锦明, 李玉生, 申琳, 曾宪铁
- YANG Jinming, LI Yusheng, SHEN Lin, ZENG Xiantie
- 骨关节炎中自噬相关基因的鉴定及其靶向中药成分的筛选
- Identification of autophagy-related genes in osteoarthritis and screening of targeted Chinese medicine components
- 天津中医药大学学报, 2025, 44(3): 208-215
- Journal of Tianjin University of Traditional Chinese Medicine, 2025, 44(3): 208-215
- http://dx.doi.org/10.11656/j.issn.1673-9043.2025.03.04
-
文章历史
收稿日期: 2024-12-22
2. 天津中医药大学第一附属医院, 天津 300193;
3. 天津医院, 天津 300211
2. First Teaching Hospital of Tianjin University of Traditional Chinese Medicine, Tianjin 300193, China;
3. Tianjin Hospital, Tianjin 300211, China
骨关节炎(OA)是世界上最常见的退行性关节疾病,影响60岁以上约10%的男性和18%的女性[1]。随着人口老龄化,OA的发病率预计将进一步增加[2]。如果控制不当,OA会导致关节衰竭,严重影响患者的日常生活,并给患者和医疗保健系统带来重大负担[3]。因此,需要更好地认识这种使人衰弱的疾病,这将有助于改善患者的预后。
自噬是真核细胞中一种高度保守的细胞稳态机制[4],与OA的发病密切相关[5]。在OA患者中,检测到了一些关键的自噬相关基因的低表达,并发现这些基因的低表达加速软骨细胞的凋亡[6],表明自噬在维持软骨细胞存活和防止关节软骨的降解中起关键作用。因此,有必要对OA中的自噬相关基因进行深入分析,这将为OA的治疗提供潜在的治疗靶点。
当前,非甾体类抗炎药广泛应用于OA的治疗,可有效缓解患者的疼痛和炎症反应。然而,长期使用非甾体类抗炎药可导致副作用,如胃肠道溃疡和出血[7-8]。因此,需要更安全和更有效的药物来治疗OA。近年来,研究人员将研究重点转向具有广泛药理作用的中药成分。中药单体是从中药中分离出来的具有特定空间结构的活性物质,越来越多地被研究人员用于疾病研究,具有广阔的应用前景[9]。因此,筛选中药中的有效成分将有助于治疗OA。
随着高通量测序技术的出现和广泛应用,基因组、转录组和蛋白质组基因组学等各种测序技术被广泛应用于OA等多因素、复杂疾病的研究。近年来,为了从海量的数据中获取关键信息并研究关键信息之间的相互作用,各种基于生物网络的集成分析技术得到了发展。常见的有基因调控网络、蛋白质相互作用网络、信号通路网络、代谢网络和基因共表达网络[10]。其中,通过加权基因共表达网络分析构建共表达网络,从而描述转录组测序数据中表达模式相似的基因模块与疾病表型之间的关系,进而筛选出疾病分子标志物或潜在的治疗靶点,越来越多地应用于复杂疾病的研究[11]。
研究旨在通过生物信息学分析鉴定OA中的自噬相关基因,并筛选能够靶向这些基因的中药成分。笔者从基因表达综合数据库获得了OA患者和健康对照的微阵列数据集,并筛选OA患者和健康对照之间的差异表达基因。随后,进行加权基因共表达网络分析,以确定与OA发病密切相关的枢纽基因模块和枢纽基因。通过取差异表达基因、枢纽基因和自噬相关基因的交集,确定了OA中的3个关键的自噬基因。通过另一个微阵列数据集GSE12021验证了这些枢纽基因的准确性。最后,利用这些枢纽基因筛选其靶向的中药成分,并进行分子对接验证以验证中药成分的准确性。本研究为OA的诊断和治疗提供了参考。
1 方法 1.1 数据下载和处理从基因表达综合据库[12]下载3个OA相关的微阵列数据集,数据集的信息见表 1。3个数据集的数据均在GPL96平台上生成,所使用的基因芯片均为Affymetrix Human Genome U133A Array。GSE55235数据集包含10个OA样本和10个健康对照样本,GSE55457数据集包含10个OA样本和10个健康对照样本,GSE12021数据集包含10个OA样本和9个健康对照样本,它们的组织来源相同,均来自于关节滑膜组织。首先,利用“limma”软件包对数据集GSE55235和GSE55457进行归一化处理,并输出标准化后的基因表达矩阵。然后通过“limma”软件包筛选OA患者和健康对照之间的差异表达基因。差异表达基因的阈值设置为|log2FC| > 0.585。为了避免假阳性结果,设置矫正后的P值< 0.05。
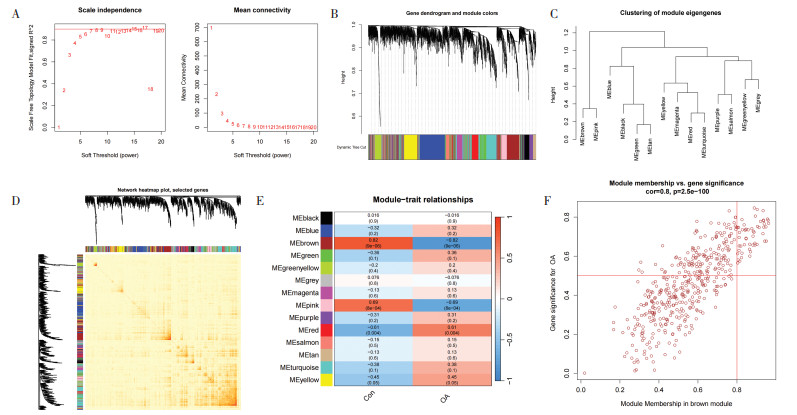
使用“WGCNA”软件包进行加权基因共表达网络分析以鉴定OA中的枢纽基因模块并筛选模块中的枢纽基因。首先,分别计算微阵列数据集GSE55235和GSE55457中每个基因表达的方差。为了确保准确性,选择方差最显著的前25%的基因进行后续分析。选择最优的软阈值并设置无标度R2为0.9以构建加权邻接矩阵,并将其转化为拓扑重叠矩阵。之后,使用基于分层聚类树算法的TOM差异度量(1-TOM)来获得基因模块(最小模块的大小设置为50)。基因模块与OA的相关性表现为模块显著性,基因与OA之间的相关性表现为基因显著性。选择与OA相关系数最高的基因模块作为枢纽基因模块,并筛选基因显著性>0.5的基因作为枢纽基因。
1.3 OA中关键自噬相关基因的鉴定从人类自噬数据库获得了222个自噬相关基因[13]。通过将差异表达基因、枢纽基因和自噬相关基因取交集,确定OA中关键自噬相关基因。
1.4 关键自噬相关基因的验证为了评估关键自噬相关基因的准确性,对验证数据集GSE12021进行受试者工作特征曲线分析。使用“pROC”软件包计算曲线下面积,曲线下面积越大表示基因区分OA患者和健康对照的准确率越高,一般认为,曲线下面积大于0.7表示基因具有良好的诊断价值,即这些基因能够很好地区分OA患者和健康对照。
1.5 筛选靶向中药成分通过CTD数据库[14]对关键的自噬相关基因进行分析,以筛选能够靶向这些基因的中药成分。CTD数据库是一个综合的基因组学数据库,被广泛用于筛选药物与基因之间的相互作用关系[14]。首先,在CTD数据库中检索与这些基因存在相互作用的化学成分,并通过数据库提供的“基因-化学成分”相互作用指数,筛选出与关键基因具有较高相互作用指数的中药成分。之后,对筛选出的中药成分进行进一步筛选,通过CTD数据库获得“基因-化学成分-疾病”三者之间的关联,在与OA的治疗具有特定关联的化学成分中筛选中药成分,并与之前获得的与关键基因具有较高相互作用指数的中药成分取交集,以获得在OA的治疗中能够靶向这些基因的中药成分。
1.6 分子对接验证为了验证所筛选的中药成分的准确性,进行了分子对接验证。首先,从RCSB PDB数据库中获得关键自噬相关基因的三维结构这些基因对应的PDB ID,见表 2。并通过PubChem数据库获得中药成分的三维结构。在进行分子对接之前,通过Chem3D软件对中药成分的三维结构进行能量最小化处理。并使用AutoDock软件将基因结构去除水分子并添加氢键,将其输出为配体;对中药成分结构进行添加氢键处理,将其输出为受体。通过AutoDock Vina软件进行分子对接模拟和结合能计算。设置每个配体与受体分别对接10次,对接过程中的其他参数均设置为默认值。一般认为,分子对接的结合能越小,化合物与靶标之间的结合力和亲和力越强,结合构象越稳定。若该值小于-5.0 kcal/mol,则认为化合物与靶标的结合良好,若小于-7.0 kcal/mol,则认为化合物与靶标的结合极强。
通过R软件(版本:4.2.2)进行统计分析。使用独立样本t检验进行组间比较,P值或矫正后的P值< 0.05则代表差异具有统计学意义。
2 结果 2.1 OA中差异表达基因的鉴定通过差异分析,在微阵列数据集GSE55235中获得了1 490个差异表达基因,在GSE55457数据集中获得了497个差异表达基因。通过火山图和热图对这些差异表达基因进行可视化,见图 1。
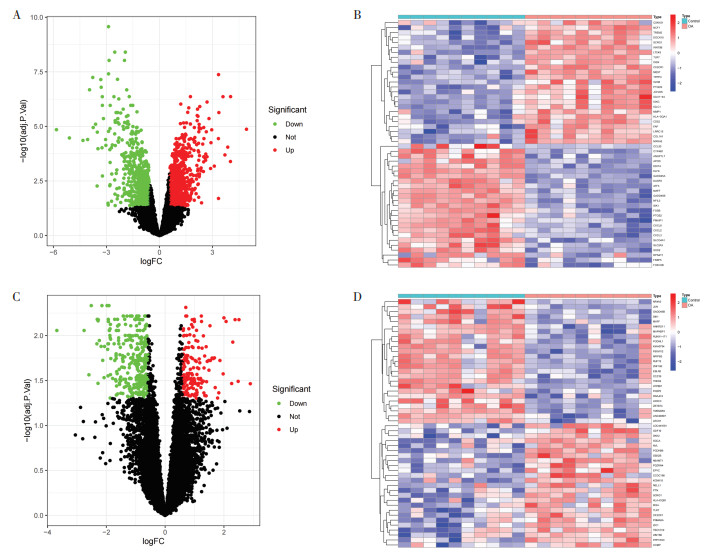
|
| 注:图A,微阵列数据集GSE55235中差异表达基因的火山图;图B,微阵列数据集GSE55235中差异表达基因的热图;图C,微阵列数据集GSE55457中差异表达基因的火山图;图D,微阵列数据集GSE55457中差异表达基因的热图。 图 1 差异表达基因的火山图和热图 |
为了获得与OA发病密切相关的枢纽基因模块并筛选出枢纽基因,笔者通过WGCNA软件包对微阵列数据集GSE55235和GSE55457分别进行分析。在微阵列数据集GSE55235中,最佳软阈值被确定为4并设置无标度R2为0.9时,确定了共表达的7个基因模块。之后选取了与OA相关性最强的基因模块(青绿色模块)作为枢纽基因模块,通过设置基因显著性>0.5,最终获得了青绿色模块中的枢纽基因,见图 2。在微阵列数据集GSE55457中,最佳软阈值被确定为6并设置无标度R2为0.9时,确定了共表达的14个基因模块。选取与OA相关性最强的棕色模块作为枢纽基因模块,并通过设置基因显著性>0.5,最终获得了棕色模块中的枢纽基因,见图 3。
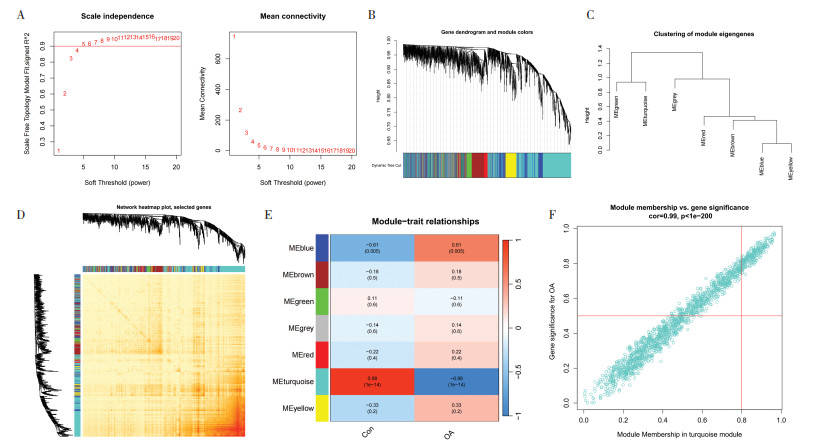
|
| 注:图A,软阈值的选择,最佳软阈值为4;图B,共表达模块的聚类树树状图,不同的颜色代表不同的共表达模块;图C,模块特征基因的聚类;图D,7个模块之间相关性的代表性热图;图E,模块特征基因与临床状态的相关性分析,每行代表一个模块,每列代表一个临床状态;图F,青绿色模块中的模块显著性与基因显著性之间的散点图。 图 2 微阵列数据集GSE55235的加权基因共表达网络分析 |
|
| 注:图A,软阈值的选择,最佳软阈值为6;图B,共表达模块的聚类树树状图,不同的颜色代表不同的共表达模块;图C,模块特征基因的聚类;图D,14个模块之间相关性的代表性热图;图E,模块特征基因与临床状态的相关性分析,每行代表一个模块,每列代表一个临床状态;图F,棕色模块中的模块显著性与基因显著性之间的散点图。 图 3 微阵列数据集GSE55457的加权基因共表达网络分析 |
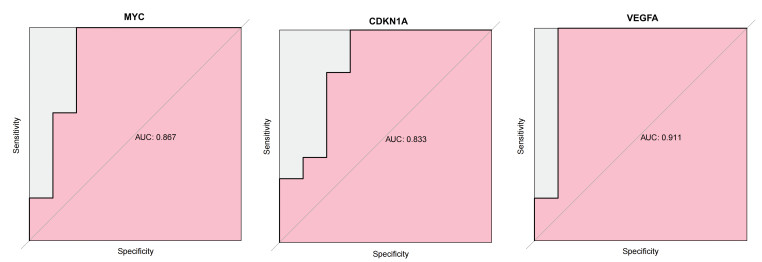
通过整合OA中的差异表达基因、枢纽基因和自噬相关基因,获得了OA中3个关键的自噬基因,见图 4。微阵列数据集GSE12021的受试者工作特征曲线显示这些关键自噬基因的曲线下面积均>0.8,见图 5,提示这3个自噬基因均具有良好的诊断能力。

|
| 图 4 差异表达基因、枢纽基因与自噬相关基因的韦恩图 |
|
| 图 5 OA中关键自噬基因的受试者工作特征曲线 |
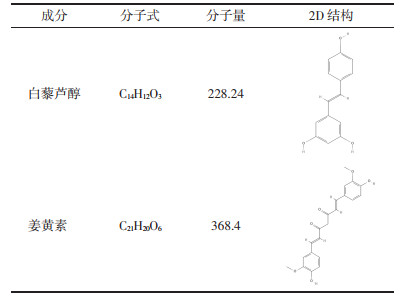
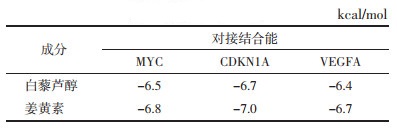
通过CTD数据库对这些关键基因的靶向中药成分进行筛选,发现白藜芦醇、槲皮素、姜黄素和丹参酮ⅡA等中药成分与这些基因具有较高的相互作用指数。通过“基因-中药成分-疾病”相互作用关系的进一步筛选,在这些中药成分中,白藜芦醇和姜黄素与这些基因及OA均具有密切关联,表明其可能是关键的中药成分,白藜芦醇和姜黄素的详细信息,见表 3。可能有助于OA的治疗。
白藜芦醇、姜黄素与MYC、CDKN1A和VEGFA分别对接10次,所有的结合自由能最低值均小于-6 kcal/mol。分子对接结果显示白藜芦醇、姜黄素和关键自噬基因之间可以形成最佳的嵌合结构,有助于OA治疗,见表 4。
OA是最常见的慢性退行性关节疾病,严重影响了患者的生活质量,由于其病因尚未完全阐明,目前的临床治疗只能提供有限的症状缓解,但不能阻止疾病进展[15]。研究表明,自噬水平的降低会加速OA和衰老相关疾病的发生;相反,激活自噬有助于减轻实验性OA的严重程度[16]。然而,自噬与OA之间的关系目前尚不完全清楚。
为了探究OA中的自噬相关基因,笔者开展了相关研究。通过分析基因表达综合数据库中公开可用的OA微阵列数据集GSE55235和GSE55457,鉴定了OA患者和健康对照之间的差异表达基因。通过加权基因表达网络分析,分别获得了与OA发病密切相关的基因模块和枢纽基因。通过将差异表达基因、枢纽基因和自噬相关基因取交集,获得了3个关键自噬基因MYC、CDKN1A和VEGFA。通过对验证数据集GSE12021进行受试者工作特征曲线分析,证实了这些关键基因的准确性(受试者工作特征曲线下面积均>0.8)。
一些研究已经发现,MYC、CDKN1A和VEGFA在OA的发病中发挥着重要作用。在人软骨细胞中检测到了CDKN1A基因的低表达[17]。进一步的研究发现,CDKN1A的低表达会抑制软骨细胞增殖,从而加速OA进程[18]。MYC通过编码转录因子来调节转录活性和细胞的增殖、生长过程,有助于维持细胞的正常功能[19]。在OA患者的关节软骨中,检测到了MYC的低表达[20],推测MYC的低表达可能影响了软骨细胞的正常功能。VEGFA又称为VEGF,在OA患者中,VEGF的表达显著高于健康对照[21]。VEGF会诱导炎症细胞因子MMP-1和MMP-3的表达,从而影响软骨细胞的正常功能,加速关节软骨的破坏[22]。此外,研究发现,抑制VEGF的表达有助于促进关节软骨的修复[23]。这些研究表明MYC、CDKN1A和VEGFA基因的表达异常会影响软骨细胞的正常功能,加速关节软骨退变,加速疾病进程。
由于中药治疗OA的历史悠久、临床使用广泛、疗效确切,且中药具有多通路、多靶点、多作用环节等优势,在OA的治疗中发挥着不可替代的作用。因此,针对这些基因筛选潜在的治疗药物,将有助于延缓OA进程。基于这3个关键的自噬基因,通过CTD数据库筛选了一些靶向的中药成分,并进行了分子对接以揭示药物与这些基因靶点之间的对接亲和力。发现白藜芦醇、姜黄素与这3个关键基因均具有良好的对接亲和力,它们可能有助于OA的治疗。
白藜芦醇是天然的酚类化合物,广泛存在于常见的食物如浆果、葡萄和桑椹中[24-26]。研究表明,白藜芦醇具有抗炎、抗凋亡和维持软骨稳态的特性,并能激活自噬[27]。而自噬水平的异常降低会加速OA进程[28],动物实验及临床前试验表明白藜芦醇可以激活自噬,延缓关节软骨退化[29-30],这表明白藜芦醇是一种有潜力的中药成分,能够激活自噬、抑制炎症和软骨细胞凋亡,有助于延缓疾病进程,促进软骨损伤的修复。姜黄素是从姜黄根茎中提取的一种多酚色素,被广泛用于缓解炎症及退行性疾病[31]。研究发现,姜黄素具有良好的抗炎和抗氧化特性,有助于延缓OA患者的关节软骨退变。此外,姜黄素还可作为线粒体自噬激活剂,维持线粒体稳态[32]。线粒体自噬是自噬的一种特殊形式,可调节功能障碍线粒体的功能,是线粒体稳态的关键机制[33]。研究发现,线粒体自噬在OA的发病机制中起着重要作用,调节线粒体自噬有助于OA的治疗[34]。因此,白藜芦醇和姜黄素是靶向调节OA自噬的潜在中药成分,有助于OA的治疗。它们能够抑制炎症细胞因子的释放并激活自噬,有助于维持关节软骨稳态。由于白藜芦醇及姜黄素与MYC、CDKN1A和VEGFA这些关键自噬基因均具有较高的对接亲和力和结合活性,推测它们将通过激活MYC和CDKN1A基因的表达并抑制VEGFA基因的表达,从而发挥激活自噬及抑制炎症的药理作用,促进软骨损伤的修复。
值得注意的是,研究也存在一些局限性。首先,笔者对之前发表的数据集进行了重新挖掘和分析。其次,仅使用一个数据集而非多个数据集进行验证,且的研究中数据集的样本量较小,这可能导致结果出现偏差。因此,需要更大的样本量来证实我们的研究结果。最后,需要进行更深入的实验研究,如qRT-PCR和western blotting等实验来探究关键自噬相关基因在OA中的作用机制,并探究白藜芦醇和姜黄素通过这些基因调控OA的具体药理作用机制。尽管如此,笔者的研究为未来开展相关研究提供了有益的参考。
| [1] |
WOOLF A D, PFLEGER B. Burden of major musculoskeletal conditions[J]. Bulletin of the World Health Organization, 2003, 81(9): 646-656. |
| [2] |
JOHNSON V L, HUNTER D J. The epidemiology of osteoarthritis[J]. Best Practice & Research Clinical Rheumatology, 2014, 28(1): 5-15. |
| [3] |
LITWIC A, EDWARDS M H, DENNISON E M, et al. Epidemiology and burden of osteoarthritis[J]. British Medical Bulletin, 2013, 105: 185-199. DOI:10.1093/bmb/lds038 |
| [4] |
CHUN Y, KIM J. Autophagy: An essential degradation program for cellular homeostasis and life[J]. Cells, 2018, 7(12): 278. DOI:10.3390/cells7120278 |
| [5] |
ROCKEL J S, KAPOOR M. Autophagy: Controlling cell fate in rheumatic diseases[J]. Nature Reviews Rheumatology, 2017, 13(3): 193. |
| [6] |
ZHANG Y, VASHEGHANI F, LI Y H, et al. Cartilage-specific deletion of mTOR upregulates autophagy and protects mice from osteoarthritis[J]. Annals of the Rheumatic Diseases, 2015, 74(7): 1432-1440. DOI:10.1136/annrheumdis-2013-204599 |
| [7] |
TARUC-UY R L, LYNCH S A. Diagnosis and treatment of osteoarthritis[J]. Primary Care, 2013, 40(4): 821-836. DOI:10.1016/j.pop.2013.08.003 |
| [8] |
RODRIGUEZ-MERCHAN E C. Topical therapies for knee osteoarthritis[J]. Postgraduate Medicine, 2018, 130(7): 607-612. DOI:10.1080/00325481.2018.1505182 |
| [9] |
ZHOU H L, MA C, WANG C, et al. Research progress in use of traditional Chinese medicine monomer for treatment of non-alcoholic fatty liver disease[J]. European Journal of Pharmacology, 2021, 898: 173976. DOI:10.1016/j.ejphar.2021.173976 |
| [10] |
GUPTA O P, DESHMUKH R, KUMAR A, et al. From gene to biomolecular networks: A review of evidences for understanding complex biological function in plants[J]. Current Opinion in Biotechnology, 2022, 74: 66-74. DOI:10.1016/j.copbio.2021.10.023 |
| [11] |
LIN W W, WANG Y X, CHEN Y S, et al. Role of calcium signaling pathway-related gene regulatory networks in ischemic stroke based on multiple WGCNA and single-cell analysis[J]. Oxidative Medicine and Cellular Longevity, 2021, 2021: 8060477. DOI:10.1155/2021/8060477 |
| [12] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: Archive for functional genomics data sets: Update[J]. Nucleic Acids Research, 2013, 41(Database issue): D991-D995. |
| [13] |
MOUSSAY E, KAOMA T, BAGINSKA J, et al. The acquisition of resistance to TNFα in breast cancer cells is associated with constitutive activation of autophagy as revealed by a transcriptome analysis using a custom microarray[J]. Autophagy, 2011, 7(7): 760-770. DOI:10.4161/auto.7.7.15454 |
| [14] |
DAVIS A P, WIEGERS T C, WIEGERS J, et al. CTD tetramers: A new online tool that computationally links curated chemicals, genes, phenotypes, and diseases to inform molecular mechanisms for environmental health[J]. Toxicological Sciences, 2023, 195(2): 155-168. DOI:10.1093/toxsci/kfad069 |
| [15] |
MARTEL-PELLETIER J, BARR A J, CICUTTINI F M, et al. Osteoarthritis[J]. Nature Reviews Disease Primers, 2016, 2: 16072. DOI:10.1038/nrdp.2016.72 |
| [16] |
CARAM?S B, HASEGAWA A, TANIGUCHI N, et al. Autophagy activation by rapamycin reduces severity of experimental osteoarthritis[J]. Annals of the Rheumatic Diseases, 2012, 71(4): 575-581. DOI:10.1136/annrheumdis-2011-200557 |
| [17] |
XIE J X, DENG Z Q, ALAHDAL M, et al. Screening and verification of hub genes involved in osteoarthritis using bioinformatics[J]. Experimental and Therapeutic Medicine, 2021, 21(4): 330. DOI:10.3892/etm.2021.9761 |
| [18] |
GANG X, XU H, SI L, et al. Treatment effect of CDKN1A on rheumatoid arthritis by mediating proliferation and invasion of fibroblast-like synoviocytes cells[J]. Clinical and Experimental Immunology, 2018, 194(2): 220-230. DOI:10.1111/cei.13161 |
| [19] |
O'DONNELL K A, WENTZEL E A, ZELLER K I, et al. C-Myc-regulated microRNAs modulate E2F1 expression[J]. Nature, 2005, 435(7043): 839-843. DOI:10.1038/nature03677 |
| [20] |
FISCH K M, GAMINI R, ALVAREZ-GARCIA O, et al. Identification of transcription factors responsible for dysregulated networks in human osteoarthritis cartilage by global gene expression analysis[J]. Osteoarthritis and Cartilage, 2018, 26(11): 1531-1538. DOI:10.1016/j.joca.2018.07.012 |
| [21] |
YUAN Q, SUN L, LI J J, et al. Elevated VEGF levels contribute to the pathogenesis of osteoarthritis[J]. BMC Musculoskeletal Disorders, 2014, 15: 437. DOI:10.1186/1471-2474-15-437 |
| [22] |
ENOMOTO H, INOKI I, KOMIYA K, et al. Vascular endothelial growth factor isoforms and their receptors are expressed in human osteoarthritic cartilage[J]. The American Journal of Pathology, 2003, 162(1): 171-181. DOI:10.1016/S0002-9440(10)63808-4 |
| [23] |
NAGAI T, SATO M, KOBAYASHI M, et al. Bevacizumab, an anti-vascular endothelial growth factor antibody, inhibits osteoarthritis[J]. Arthritis Research & Therapy, 2014, 16(5): 427. |
| [24] |
BURNS J, YOKOTA T, ASHIHARA H, et al. Plant foods and herbal sources of resveratrol[J]. Journal of Agricultural and Food Chemistry, 2002, 50(11): 3337-3340. DOI:10.1021/jf0112973 |
| [25] |
STERVBO U, VANG O, BONNESEN C. A review of the content of the putative chemopreventive phytoalexin resveratrol in red wine[J]. Food Chemistry, 2007, 101(2): 449-457. DOI:10.1016/j.foodchem.2006.01.047 |
| [26] |
SALES J M, RESURRECCION A. Resveratrol in peanuts[J]. Critical Reviews in Food Science and Nutrition, 2023, 54: 734-770. |
| [27] |
YANG S L, SUN M L, ZHANG X N. Protective effect of resveratrol on knee osteoarthritis and its molecular mechanisms: A recent review in preclinical and clinical trials[J]. Frontiers in Pharmacology, 2022, 13: 921003. |
| [28] |
JEON H, IM G I. Autophagy in osteoarthritis[J]. Connective Tissue Research, 2017, 58(6): 497-508. |
| [29] |
QIN N, WEI L W, LI W Y, et al. Local intra-articular injection of resveratrol delays cartilage degeneration in C57BL/6 mice by inducing autophagy via AMPK/mTOR pathway[J]. Journal of Pharmacological Sciences, 2017, 134(3): 166-174. |
| [30] |
YI H, ZHANG W, CUI Z M, et al. Resveratrol alleviates the interleukin-1β-induced chondrocytes injury through the NF-κB signaling pathway[J]. Journal of Orthopaedic Surgery and Research, 2020, 15(1): 424. |
| [31] |
BANNURU R R, OSANI M C, AL-EID F, et al. Efficacy of curcumin and Boswellia for knee osteoarthritis: Systematic review and meta-analysis[J]. Seminars in Arthritis and Rheu- matism, 2018, 48(3): 416-429. |
| [32] |
JIN Z Z, CHANG B H, WEI Y L, et al. Curcumin exerts chondroprotective effects against osteoarthritis by promoting AMPK/PINK1/Parkin-mediated mitophagy[J]. Biomedicine & Pharmacotherapy, 2022, 151: 113092. |
| [33] |
PALIKARAS K, LIONAKI E, TAVERNARAKIS N. Mechanisms of mitophagy in cellular homeostasis, physiology and pathology[J]. Nature Cell Biology, 2018, 20(9): 1013-1022. |
| [34] |
SUN K, JING X Z, GUO J C, et al. Mitophagy in degenerative joint diseases[J]. Autophagy, 2021, 17(9): 2082-2092. |
 2025, Vol. 44
2025, Vol. 44